Hvad kan DNA fortælle om os?
Alt levende indeholder DNA, inklusiv os mennesker. Du har sikkert allerede lært, at DNA er koden for proteiner i vores krop. Men hvor meget information kan man faktisk få om et menneske, hvis man kigger på personens DNA? Og hvad kan denne information bruges til?
I temaet ‘Information I DNA’ skal du undersøge alt dette ved at se på, hvordan man bruger DNA til retsgenetiske undersøgelser. I forløbet skal du hjælpe politiet med at identificere personer ud fra deres DNA og samtidig lære om, hvor meget dit DNA faktisk kan afsløre om dig og din familie. Endelig skal du lære om, hvilke fordele og ulemper der er ved at lave store DNA databaser og tage stilling til de etiske problemstillinger, de medfører.
Velkommen til undervisningsmaterialet ‘Information i DNA’.
Følgende delmål af de faglige mål i læreplanen for Bioteknologi A dækkes i forløbet ’Information i DNA’:
- tilrettelægge og udføre eksperimenter og undersøgelser under hensyntagen til laboratoriesikkerhed og risikomomenter ved arbejde med biologisk materiale (modul 0+1)
- bearbejde data fra kvalitative og kvantitative eksperimenter og undersøgelser og dokumentere eksperimentelt arbejde hensigtsmæssigt (modul 0 + 1)
- anvende fagbegreber, fagsprog, relevante repræsentationer og modeller til beskrivelse og forklaring af iagttagelser og til analyse af bioteknologiske problemstillinger (3+5+6+7)
- demonstrere viden om fagets identitet og metoder (især modul 2 + 3 + 4 + 6)
- anvende relevante matematiske repræsentationer, modeller og metoder til analyse og vurdering (modul 5 + 6)
- anvende digitale værktøjer, herunder fagspecifikke og matematiske, i en konkret faglig sammenhæng (modul 5 + 6)
- anvende fagets viden og metoder til vurdering og perspektivering i forbindelse med samfundsmæssige, teknologiske, miljømæssige og etiske problemstillinger med bioteknologisk indhold og til at udvikle og vurdere løsninger (modul 8)
Kernestof, der dækkes i forløbet:
- genetik og molekylærbiologi: nedarvningsprincipper, meiose, genregulering, mutation, genteknologi, anvendt bioinformatik
- eksperimentelle metoder: PCR, elektroforese
Supplerende stof, der dækkes i forløbet:
- ny forskning og nye bioteknologiske metoder
- bioetik
Der skal indgå områder med relation til elevernes hverdag og samfundsmæssige, teknologiske eller miljømæssige problemstillinger med bioteknologisk indhold.
Der skal indgå materiale på engelsk samt, når det er muligt, på andre fremmedsprog.
Modul 0: Blodsporet
I dette modul skal du:
- Have en kort, generel introduktion til forløbet.
- Skrive ned, hvad du ved og hvad du tror, at DNA kan fortælle om en person.
- Lære om, hvordan DNA-spor bliver fundet på et gerningssted.
- Designe et forsøg, der kan undersøge, hvor små mængder blod man kan opspore med Luminol testen.
I dette forløb skal du undersøge, hvilke oplysninger man kan finde frem til ved at undersøge DNA. Inden du går i gang, skal du bruge 5 minutter på at lave en brainstorm med udgangspunkt i din forhåndsviden om DNA. Hvad ved du, at DNA bruges til? Og hvad tror du, at DNA kan bruges til? Skriv ned hvad du ved eller tror det bruges til.
Når vi har været igennem forløbet, vender vi tilbage og ser, om det du troede nu også er tæt på virkeligheden.
“Morder fældet af DNA-bevis. Offer identificeret pga. DNA-rester.” Den sætning har du sikkert læst i nyhederne eller set i din yndlings TV-serie. Men hvad er et DNA-spor egentlig, og hvordan findes de på et gerningssted? Snak sammen med din makker og sæt ord på, hvad der skal til for, at noget er et DNA-spor, hvad du tror er de mest almindelige DNA-spor, og hvordan de kan findes på et gerningssted.
Det første step i alle undersøgelser af DNA er at få fat i en DNA-prøve. Nogle gange er det nemt, hvis en person er under mistanke og personen er indforstået med at afgive en DNA-prøve. DNA kan således indsamles via et kindskrab af celler fra slimhinden i munden.
Men ofte har politiet ingen mistænkt. Her skal DNA-sporet findes på gerningsstedet, hvor politiets retsteknikere prøver at finde DNA i rester af blod, spyt, hår eller sæd eller ander væv. Da DNA kan udvindes fra meget små prøver, er det langt fra altid, at DNA-spor er synlige med det blotte øje.
Du skal derfor i næste modul undersøge Luminol testen, en test, der bruges til identifikation af blodspor. I din gruppe skal du nu designe 2 forsøg, der undersøger, hvor følsom testen er i forhold til at identificere blodspor, og om der kan komme falsk positive svar.
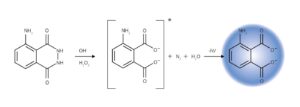
En meget brugt standard test til identifikation af blodspor er Luminol testen. Luminol er et stof, der under de rette reaktionsforhold kan indgå i en reaktion, der laver kemoluminiscens. Kemoluminiscens er, når der udsendes lys under en kemisk reaktion under moderat varmeudvikling.
Luminol kan under basiske forhold oxideres af et oxidationsmiddel som hydrogenperoxid \(H_2O_2\). Reaktionen kræver dog tilstedeværelsen af en katalysator, som f.eks jern-ioner. Reaktionen danner et ustabilt mellemprodukt med en exciteret elektron, der når den falder tilbage mod en bane tættere på kernen afgiver energi, der udsendes som en blåt lysende foton (lyspartikel).
Men hvorfor kan reaktionen bruges til at finde blodspor? Det skal du undersøge nu sammen med din gruppe ved at bruge din bioteknologi grundbog eller ved at søge på nettet og undersøge, hvad blod består af. Brug de tre hjælpespørgsmål nederst som vejledning til, hvad du skal finde ud af.
- Hvilke celletyper findes der i blod?
- Hvilke celler indeholder jern-ioner, der kan katalysere Luminol reaktionen?
- Hvilke celler i blodet kan bruges til at udvinde DNA fra?
Sammen med din gruppe skal du i næste modul teste, hvor små mængder af blod det er muligt at spore ved brug af Luminol testen samt om andre ting kan udløse reaktionen. Når I designer jeres forsøg skal I skrive ned, hvordan I vil lave jeres undersøgelser. I skal bruge følgende fagtermer i jeres beskrivelse:
- Uafhængig variabel: Det som I ændrer I jeres forsøg.
- Afhængig variabel: Det som I observerer sker i forsøget.
- Kontrolforsøg: Forsøg, hvor der ikke er foretaget nogen ændring.
- Variabel kontrol: Andre ting der skal holdes konstant under forsøget, for at I kan sammenligne resultaterne.
- Replikater: Er det muligt for jer at lave den samme test mere end en gang?
Til delforsøg 1 vil I have følgende til rådighed:
- Sprayflaske med Luminol opløsning (udleveres af jeres lærer når I er klar med resten)
- Filtrer papir
- Micropipette (10 µL og 200 µL) plus pipettespidser
- Fingerprikker (til at skaffe lidt blod) + desinfektionsservietter.
- 0,5 mL Eppendorfrør
- Pincet
- Diverse glasvarer i laboratoriet i laboratoriet + demineraliseret vand.
- Blyant + spritpen
Et godt startvolumen blod at undersøge er 10 µL – det skaffes ved, at en I jeres gruppe bliver prikket i fingeren med en fingerprikker. Bloddråben kan så suges op med en 10 µL pipette.
Til delforsøg 2 har i ydermere følgende til rådighed:
Klorin, opvaskemiddel, rustent søm, ketchup, revet peberrod, rød saftevand.
Skriv jeres forsøgsdesign ind i journalen, der kan downloades her på siden.
Modul 1: Luminol test og DNA oprensning
I dette modul skal du:
- Undersøge Luminol testen med de to forsøg, som jeres gruppe har designet.
- Sammenligne jeres testresultater med to andre tests, som kan identificere blodspor og derefter vurdere fordele og ulemper ved de tre metoder.
- Lære om en metode til at oprense DNA vha. magnetiske kugler.
Din gruppe har nu undersøgt Luminol testen. Skriv jeres svar ned, så vi kan sammenligne klassens resultater.

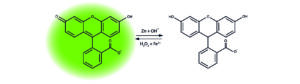
Fluorescein testen er endnu en test, der kan bruges til at finde blodspor. Fluorescein er – som navnet også angiver – et stærkt fluorescerende stof. Fluorescens er, når elektroner i et stof exciteres og straks ved henfald til en bane tættere på kernen vil udsende lys med en bølgelængde, der vil være længere end det lys, det blev bestrålet med. Når fluorescein exciteres med enten blåt eller ultraviolet lys, vil det udsende et kraftigt grønt lys.
Fluorescein kan omdannes til en reduceret farveløs form ved en reduktion med zink under basiske forhold. Udsættes den reducerede form for et stærkt oxidationsmiddel som hydrogenperoxid, vil det kunne oxideres tilbage til den fluorescerende form. Det kræver dog tilstedeværelsen af en katalysator såsom jernioner, og derfor kan fluorescein også bruges til at identificere blodspor.
Ved forsøg har det vist sig, at fluorescein kan registrere blod, der er fortyndet ca. 1:100.000. Det giver ikke falsk positive resultater ved reaktion med klorin (hypochlorit) og betragtes generelt som værende mindre giftigt end Luminol.
 Kastle-Meyer testen er endnu en test, der bruges til at finde blodspor. Til testen bruger man pH indikatoren phenolphthalein. Phenolphthalein er farveløs ved pH værdier under 8, men kraftig pink under mere basiske forhold.
Kastle-Meyer testen er endnu en test, der bruges til at finde blodspor. Til testen bruger man pH indikatoren phenolphthalein. Phenolphthalein er farveløs ved pH værdier under 8, men kraftig pink under mere basiske forhold.
Phenolphthalein kan dog også gøres farveløs under basiske forhold, hvis molekylet ligesom fluorescein reduceres ved hjælp af zink i basisk opløsning. Ligesom fluorescein kan det bringes tilbage til sin farvede form, hvis det oxideres af et stærkt oxidationsmiddel såsom hydrogenperoxid under tilstedeværelse af en katalysator såsom jernioner.
Kastle-Meyer testen kan bruges til at spore blod, der er fortyndet til ca. 1:20.000. Stoffet laver de samme falsk positive resultater som Luminol testen. Stoffet er mindre giftigt end luminol, men har nogenlunde samme giftighed som fluorescein.

I har nu lavet jeres forsøg med Luminol testen og læst om Fluorescein testen og Kastle-Meyer testen. Alle tre tests bruges til at identificere blodspor. Men hvorfor er det ikke nok med kun en test? Det skal I prøve at finde ud af nu.
I skal vurdere, hvilke fordele og hvilke ulemper hver type test har. Undersøg, hvordan hver type test udføres, og hvad der skal til før, at det er muligt at se et DNA-spor. Er der nogle gerningssteder, hvor en test vil være svær at udføre? Sammenlign også følsomhed og giftighed af de tre tests.
Skriv jeres overvejelser ned som gruppe.

Når et DNA-spor er fundet, så er første trin i processen at oprense DNA’et fra cellerne. Celler indeholder DNA’ase enzymer, der nedbryder DNA samt en lang række proteiner, der binder sig til DNA’et. Det er derfor meget vigtigt at få fjernet alt andet end det rene genomiske DNA fra prøverne.
Der findes flere forskellige metoder til at oprense DNA. En ofte anvendt metode er, hvor man bruger magnetiske kugler til oprensningen, på engelsk kaldet “magnetic beads”. Metoden udnytter, at man kan styre, hvor magnetiske kuglerne er ved at sænke elektrisk ledende metalstænger ned i opløsning af de magnetiske kugler og DNA.
Eftersom DNA har negativt ladede phosphatgrupper, er de hydrofile og vil meget gerne være i en vandig opløsning. Men hvis man tilsætter stoffer til vandet, der forhindrer, at der dannes hydrogenbindinger mellem vandmolekylerne, vil DNA’et kunne tiltrækkes til de magnetiske kugler. Ved at holde kuglerne fast med elektromagneter kan man fjerne alt væske omkring dem og på den måde fjerne uønskede enzymer og proteiner.
Du skal nu se en animation af, hvordan en DNA-oprensning med magnetiske kugler foregår. Beskriv, hvad der sker i de tre faser, der vises i animationen. Noter også, hvilke fordele der nævnes ved metoden.
Modul 2: PCR af DNA-spor
I dette modul skal du:
- Lære om, hvad Short Tandem Repeats er, og hvordan de bruges til at lave DNA-profiler.
- Forklare og forstå, hvad PCR-reaktioner er ud fra 3 animationer.
- Designe PCR-primere til D10S1248 DNA-området, der bruges til at lave DNA-profiler.
Når DNA’et fra DNA-sporet er fundet og oprenset, er det klar til at blive analyseret. Hvordan det analyseres afhænger af, hvad man ønsker at finde ud af om personen. Det er det, som du skal lære om i resten af forløbet.
Den mest anvendte analyse er at bruge DNA-sporet som en måde at identificere en person på. Siden slutningen af 1980’erne har man brugt DNA til at lave DNA-profiler i Danmark. Metoden er et meget stærkt redskab i forhold til at fastslå om en person har været til stede på et gerningssted. Metoden har desuden hjulpet med at opklare en række mord og voldtægtssager, som ikke kunne opklares tidligere.
I dette modul skal du først lære om, hvordan DNA kan opformeres ved PCR-metoden, så der bliver nok DNA til at lave en analyse. For at kunne lave PCR skal der designes PCR-primere. Dette skal du prøve at gøre i dette modul. I næste modul skal du lære om, hvordan DNA’et analyseres og prøve at løse 3 forskellige cases ved at analysere DNA-profiler.
Se videoen som forklarer, hvad Short Tandem Repeat områder er, og hvorfor de kan bruges til at identificere forskellige mennesker. Besvar de 4 nedenstående forståelsesspørgsmål inden du kommer til modulet.
- Hvad er en Short Tandem repeat?
- Hvor finder man områder med Short Tandem Repeats?
- Forklar, hvorfor man altid har to kopier af et Short Tandem Repeat område.
- Overvej, hvorfor det ville være mere sikkert at lave DNA sammenligninger af 21 STR-områder fremfor 17.
 Hvilke basekombinationer og hvor mange gentagelser, der er, varierer fra STR-område til STR-område. På tabellen til højre ses variationen i de 17 områder som blev analyseret frem til 2021 i Danmark. Som tabellen viser, er det oftest kombinationer af 4 basepar, der gentages, men der er afvigelser – enten ved, at det kun er 3 basepar, eller at der sker et baseskift i gentagelserne undervejs.
Hvilke basekombinationer og hvor mange gentagelser, der er, varierer fra STR-område til STR-område. På tabellen til højre ses variationen i de 17 områder som blev analyseret frem til 2021 i Danmark. Som tabellen viser, er det oftest kombinationer af 4 basepar, der gentages, men der er afvigelser – enten ved, at det kun er 3 basepar, eller at der sker et baseskift i gentagelserne undervejs.
Hvis du ser på variationen, kan du få øje på, at der ved nogle STR-områder står decimaltal. For eksempel står der ud for D19S433, at det mindste antal af STR-repeats er 5,2. Det virker umiddelbart underligt, da man jo ikke kan tage 1/5 af 4 DNA-baser ud af en gentagelse.
 Forklaringen er, at STR-gentagelserne i nogle STR-områder afsluttes midt i en gentagelse og ikke efter, at den er færdig. På figuren til højre ses et eksempel, hvor en person har en almindelig STR med 6 gentagelser, en anden STR med 4 gentagelser og 2 ud af 3 basepar af endnu en gentagelse. Det skrives som 4,2, hvor ,2 viser, at der er 2 ekstra basepar ud over de 4 fulde STR gentagelser.
Forklaringen er, at STR-gentagelserne i nogle STR-områder afsluttes midt i en gentagelse og ikke efter, at den er færdig. På figuren til højre ses et eksempel, hvor en person har en almindelig STR med 6 gentagelser, en anden STR med 4 gentagelser og 2 ud af 3 basepar af endnu en gentagelse. Det skrives som 4,2, hvor ,2 viser, at der er 2 ekstra basepar ud over de 4 fulde STR gentagelser.
Modulet kan begynde med en gennemgang af lektien om STR til modulet. Snak om:
Hvad er en Short Tandem repeat?
- Hvor finder man områder med Short Tandem Repeats?
- Forklar, hvorfor man altid har to kopier af et Short Tandem Repeats’ områder.
- Overvej, hvorfor det ville være mere sikkert at lave DNA sammenligninger af 21 STR-områder fremfor 17.
Du har sikret et DNA-spor og oprenset det, men du er endnu ikke parat til at analysere DNA’et. De mængder af DNA-områder, du vil undersøge, er nemlig så små, at det er nødvendigt at mangedoble mængden af DNA først. Det gør man ved brug af en metode, der på engelsk hedder Polymerase Chain Reaction, forkortet PCR.
PCR-metoden kopierer DNA ved at efterligne en proces i vores celler. Hver gang en celle deler sig i vores krop, skal DNA’et i cellen også kopieres, hvilket gøres ved processen DNA-replikation. DNA’et kopieres ved, at enzymet DNA-polymerase bruger hver enkelt streng af et DNA-molekyle som skabelon til at lave den modsatte streng.
Du skal nu se 3 korte animationer om PCR-processen. Animationerne er uden lyd, og det er dig og din makkers opgave at forklare, hvad der sker i hver animation til hinanden. Brug din lærebog til at finde information om PCR-reaktioner. Brug de vejledende spørgsmål under hver animation som hjælp til, hvad du skal komme ind på.
Besvar følgende spørgsmål om PCR-reaktionen. Husk, at du kan stoppe animationen, når du har brug for det. Du kan til fordel se videoen på fuld skærm for at se detaljer i animationen.
- Hvad er en primer, og hvorfor binder den sig til bestemte DNA-områder?
- Hvorfor bruger man to primere?
- Hvad er funktionen af DNA-polymerasen?
- Hvad skal DNA-polymerasen bruge for at kunne lave en kopi af DNA-strengen?
- Hvorfor skal temperaturen varieres, når man laver PCR-reaktionen?
I PCR-animation 1 blev du præsenteret for de første to PCR-cyklusser. I PCR-animation 2 skal du nu se på, hvordan PCR-cyklus 3 foregår og prøve at regne ud, hvad der sker i PCR-cyklus 4.
- Hvor mange lange, mellemlange og korte DNA fragmenter er der efter 2 PCR-cyklusser?
- Hvor mange lange, mellemlange og korte DNA fragmenter er der efter 3 PCR-cyklusser?
- Hvor mange lange, mellemlange og korte DNA fragmenter vil der være efter 4 PCR-cyklusser? Begrund dit svar.
PCR-animation 3 viser antallet af DNA fragmenter, der dannes i de første 5 PCR cykler. En PCR-reaktion vil typisk blive gentaget mellem 25-35 gange.
- Hvor mange DNA-fragmenter er der dannet efter 3, 4 og 5 PCR-cykler?
- Hvad kalder man den type vækst matematisk?
- Formlen for væksten i antallet af DNA-fragmenter kan skrives som \(2^x\) hvor x=antal PCR-cykler. Hvor mange kopier vil der være teoretisk dannes af DNA-fragmenterne, hvis du laver en PCR-reaktion med 30 cykler?
Lad evt. eleverne speake til de 3 animationer på tavlen – kan vises via Powerpointen.
Spørg evt. eleverne om, hvorfor det er vigtigt at designe primere for at kunne opformere bestemte DNA-områder som overgang til øvelsen om primer design.
PCR primere er korte enkeltstrengede DNA-stykker, der binder til 3′ enden af hver af de DNA-strenge, som det dobbeltstrengede DNA består af. Herved kan DNA-polymeraser sætte sig fast på DNA-strengen og bygge en komplementær DNA-streng op af DNA-nucleotider i 5′ til 3′ retningen. Der skal bruges to primere til en PCR-reaktion: En forward primer, der binder til skabelonstrengen og en reverse primer, der binder til den kodende streng.

Primerne er det der afgør, hvilken del af det genomiske DNA, der kopieres ved PCR-reaktionen. Det er derfor vigtigt, at de specifikt binder til områderne lige før og efter DNA-stykket, der skal kopieres, så det kun er dette stykke DNA, der mangedobles. Derfor skal primerne designes, så det sikres, at de er specifikke og ikke binder til andre DNA-områder.
Rent praktisk har det også vist sig, at der er andre regler, som primere skal opfylde, for at en PCR-reaktion forløber uden problemer. Det er derfor vigtigt at kontrollere, om den designede forward og reverse primer opfylder kravene inden der laves en PCR-reaktion. Du skal nu prøve at designe et primer par, der kan bruges til at opformere STR-området D10S1248
 PCR primere skal opfylde følgende regler:
PCR primere skal opfylde følgende regler:
- De skal være mellem 18-25 basepar lange.
- De skal have et smeltepunkt mellem 52°C og 58°C.
- De skal have en såkaldt G/C clamp, hvilket betyder, at 2 eller 3 af de sidste 5 baser skal være enten C eller G.
- Den enkelte primer må ikke kunne binde til sig selv – det kaldes, at den laver primer dimer.
- Forward og reverse primeren må ikke kunne binde til hinanden – det kaldes, at de laver heterodimer.
Reglerne kan virke forvirrende, men de er alle lavet for at få PCR-reaktionen til at forløbe uden problemer. Lad os se på hvorfor.
Regel 1: En primer skal være mellem 18-25 basepar lang. Hvis en primer er under 18 basepar lang, vil det dobbeltstrengede DNA, som den kan danne, ikke være langt nok til, at DNA-polymerasen kan binde sig. Hvis den er over 25 basepar lang, er der risiko for, at primeren kan binde delvist andre steder i DNA’et end der, hvor man ønsker at give et uønsket DNA-produkt.
Regel 2: En primers smeltepunkt siger noget om, hvor godt den vil binde sig til DNA’et. Hvis smeltepunktet er for højt, risikerer primeren at binde uønskede steder i det genomiske DNA. Hvis smeltepunktet er for lavt, vil primeren binde for dårligt til DNA’et. Smeltepunktet bestemmes af antallet af C/G nucleotider i forhold til antal A/T nucleotider. Jo flere C/G nucleotider en primer indeholder, jo højere smeltepunkt vil den have grundet, at der er tre hydrogenbindinger mellem C og G, men kun to mellem A og T.
Regel 3: To eller tre ud af de sidste 5 basepar i primeren skal være G eller C, da de sikrer, at enden af primeren binder stabilt til DNA’et.
Regel 4: En primer må ikke kunne binde til sig selv, da den så ikke vil binde til DNA’et. På figuren er der vist en primer dimer, hvor alle baserne kan binde sig til hinanden. Men det er ofte nok, at der er 6-7 basepar efter hinanden, der kan binde sig, for at der dannes primer dimer.
Regel 5: Forward og reverse primerne må heller ikke kunne binde sig til hinanden. På figuren er der vist en heterodimer, hvor alle 20 basepar matcher hinanden, men igen vil det typisk være nok med 6-7 basepar efter hinanden, for at der kan dannes heterodimere.
Du skal nu bruge reglerne til at designe en forward og reverse primer ud fra følgende STR DNA-fragment,
der kommer fra det D10S1248 området på kromosom nummer 10:
|
Marker DNA-sekvensen og kopier den over i primer design vinduet. Design og test dine primere og upload
det færdige primersæt, som du har designet, som en tekstfil nederst på siden.
https://pcr-test.dataekspeditioner.dk
PCR-primere regler:
- De skal være mellem 18-25 basepar lange.
- De skal have et smeltepunkt mellem 52°C og 58°C.
- De skal have en såkaldt G/C clamp, hvilket betyder, at 2 eller 3 af de sidste 5 baser skal være enten C
eller G. - Den enkelte primer må ikke kunne binde til sig selv – det kaldes, at den laver primer dimer.
- Forward og reverse primeren må ikke kunne binde til hinanden – det kaldes, at de laver heterodimer.
Brug din nye viden til at designe endnu en forward og reverse primer til D16S539 området på kromosom
nummer 16:
|
https://pcr-test.dataekspeditioner.dk
PCR primere regler:
- De skal være mellem 18-25 basepar lange.
- De skal have et smeltepunkt mellem 52°C og 58°C.
- De skal have en såkaldt G/C clamp, hvilket er at 2 eller 3 af de sidste 5 baser skal være enten C eller G.
- Den enkelte primer må ikke kunne binde til sig selv – det kaldes, at den laver primer dimer.
- Forward og reverse primeren må ikke kunne binde til hinanden – det kaldes, at de laver heterodimer.
Modul 3: DNA profiler
I dette modul skal du:
- Lære om, hvordan en DNA-profil laves ved hjælp af kapillær gelelektroforese.
- Forstå, hvordan DNA-profiler i Danmark er opbygget, og hvordan kønsmarkøren Ameloginin virker.
- Udregne sandsynligheden for, at en person har en bestemt DNA-profil.
- Hjælpe myndighederne med at analysere DNA-profiler fra 3 forskellige cases.
Denne animation skal give din en forståelse af, hvordan PCR-fragmenter med forskellige antal Short Tandem Repeats kan adskilles ved hjælp af en metode, der hedder kapillær gelelektroforese. Du skal bruge den viden, du får fra animationen, når du i timen skal analysere din første DNA-profil.
 Det er vigtigt for efterforskere at vide, om det DNA-spor, de analyserer, stammer fra en kvinde eller en mand. Det er muligt at lave et kort over en persons kromosomer, en såkaldt karyotype, og se, om personen har to X kromosomer eller et X og et Y kromosom. Det kræver dog en ekstra analyse, hvilket gør det besværligt.
Det er vigtigt for efterforskere at vide, om det DNA-spor, de analyserer, stammer fra en kvinde eller en mand. Det er muligt at lave et kort over en persons kromosomer, en såkaldt karyotype, og se, om personen har to X kromosomer eller et X og et Y kromosom. Det kræver dog en ekstra analyse, hvilket gør det besværligt.
Forskere har derfor ledt efter et DNA-område, der er forskelligt på X og Y kromosomer. Det område, de har fundet, er på amelogenin genet, hvor der er en deletion af 6 basepar i intron 1 i den version af genet, der findes på X-kromosomet. Amelogenin genet findes også på Y-kromosomet, men her har intron 1 ikke deletionen. Genet er derfor 6 basepar længere.
Fordi der er forskel på længden af amelogenin genet alt efter, om det stammer fra et X eller et Y kromosom, kan det ligesom STR-områder adskilles ved opformering ved PCR og efterfølgende kapillær gelelektroforese. DNA-fragmentet, der opformeres, er enten 102 eller 108 basepar langt. På figuren til højre ses de to mulige udfald af analysen. Er det A eller B, der stammer fra en person, der biologisk er en mand?
Efter introduktion til modulet kan det være en fordel med en gennemgang af, hvordan kapillær gelelektroforese foregår. Få dernæst eleverne til at forklare hinanden, hvordan biologisk køn kan adskilles via Amelloginin (AMEL) markøren. Figurer til gennemgangen findes i Powerpointen til modul 3.
Nedenstående billede illustrerer, hvordan resultatet af en DNA-profil ser ud. Du skal nu prøve at analysere DNA-profilen og se, hvilken information du kan få ud af den. For at hjælpe dig på vej er der en række forståelsesspørgsmål, som du skal svare på under DNA-profilen.

- Hvor mange STR-områder analyseres i denne DNA-profil, hvis du ikke tæller kønsmarkøren med?
- Hvilket område er kønsmarkøren?
- Er DNA-profilen af en biologisk kønnet mand eller kvinde?
- Er der det samme antal STR-gentagelser i STR-område D22S1045 eller et forskelligt antal?
- Er der det samme antal STR-gentagelser i STR-området D1S1656 eller et forskelligt antal?
- For STR-område D10S1248 gælder den følgende sammenhæng mellem antallet af STR-repeats og PCR fragment længden (vist i tabellen nedenfor). Se på DNA-profilen og bestem, hvor mange STR-gentagelser der er for D10S1248 områdets to alleler.
| Antal STR-repeats | PCR fragment længde |
|---|---|
| 10 | 87 |
| 11 | 91 |
| 12 | 95 |
| 13 | 99 |
| 14 | 103 |
| 15 | 107 |
| 16 | 111 |
| 17 | 115 |
| 18 | 119 |
DNA-profiler bruges til at skelne mellem personer i retssager. De er ofte meget afgørende for, om en person kan dømmes for en alvorlig forbrydelse, der kan give mange år i fængsel, eller ej. Derfor er det ekstremt vigtigt, at sandsynligheden for, at to personer har den samme DNA-profil bliver så lille som overhovedet muligt.
For at vurdere sandsynligheden for, at to personer har de samme antal STR-gentagelser fra et STR-område, har forskere kortlagt hyppigheden af alle STR-områdernes alleler for forskellige befolkningsgrupper. Tabellen nedenfor viser hyppigheden af forskellige alleler for STR-området D10S1248 i en etnisk dansk befolkning.
| STR allel | Hyppighed |
|---|---|
| 9 | 0 |
| 10 | 0 |
| 11 | 0,003 |
| 12 | 0,032 |
| 13 | 0,311 |
| 14 | 0,309 |
| 15 | 0,181 |
| 16 | 0,126 |
For at finde sandsynligheden for en STR-allel kombination ganges sandsynligheden for den første allel med sandsynligheden for den anden allel. Ved analysen af DNA-profilen bestemte du antal STR-gentagelser fra D10S1248 til 13 og 16 gentagelser. Sandsynligheden for at have lige denne kombination i en etnisk dansk befolkning er \(0,309*0,126=0,0389\) \(0,311*0,126=0,392\) eller ca. 3,9%.
En DNA-profil udregnes ved at gange alle STR-allelers hyppighed sammen. Kønsmarkøren ameloginin er et specielt tilfælde, da en biologisk kvinde altid har XX og en biologisk mand XY. Derfor er det kun 1 allel, der varierer, og da hyppigheden af mænd og kvinder næsten er ens, sættes hyppigheden for allel 2 som 0,5.
Du skal nu prøve at udregne sandsynligheden for, at en person har den samme DNA-profil som den, du lige har analyseret. Nedenfor ses en tabel, hvor antal STR-gentagelser for hvert STR-område er angivet sammen med sandsynligheden for den fundne STR-allel.
| STR område | Allel 1 | Hyppighed | Allel 2 | Hyppighed |
|---|---|---|---|---|
| D10S1248 | 13 | 0,309 | 16 | 0,126 |
| vWA | 18 | 0,196 | 18 | 0,196 |
| D16S539 | 10 | 0,06 | 11 | 0,31 |
| D2S1338 | 20 | 0,148 | 23 | 0,105 |
| Ameloginin | 102 (X) | 1 | 102 (X) | 0,5 |
| D8S1179 | 10 | 0,102 | 13 | 0,307 |
| D21S11 | 30 | 0,26 | 30 | 0,26 |
| D18S51 | 18 | 0,076 | 20 | 0,018 |
| D22S1045 | 13 | 0,006 | 18 | 0,005 |
| D19S433 | 12 | 0,079 | 13 | 0,242 |
| TH01 | 9 | 0,146 | 9 | 0,146 |
| FGA | 19 | 0,061 | 25 | 0,084 |
| D2S441 | 14 | 0,288 | 15 | 0,052 |
| D3S1358 | 15 | 0,268 | 16 | 0,243 |
| D1S1656 | 15 | 0,129 | 15 | 0,129 |
| D12S391 | 16 | 0,027 | 22 | 0,113 |
| SE33 | 18 | 0,08 | 28,2 | 0,056 |
Kopier først data fra tabellen over i et regneark. Du skal nu udregne den samlede sandsynlighed for, at en person lige har den viste DNA-profil. Stil data logisk op i regnearket og brug dem til at lave udregningen. Når du er færdig, skal du uploade det færdige regneark med dit resultat.
Du har nu prøvet at udregne sandsynligheden for, at en person har en bestemt DNA-profil, og som du så, er sandsynligheden ekstremt lav. Selv om du kun bruger halvdelen af STR-områderne til at lave en DNA-profil, er sandsynligheden stadig meget lav. Det vil være mere end rigeligt til at løse en sag i langt de fleste tilfælde. I de næste tre opgaver skal du derfor kun analysere på de første 7 STR-områder samt Ameloginin genet, da det gør opgaverne mere overskuelige.
Din opgave er at hjælpe politiet med at finde beviser, der kan hjælpe med at opklare 3 cases, nemlig en indbrudscase, en drug rape case og en faderskabssag. Selvom der bruges DNA-profiler i alle 3 cases, er analysen af DNA-profilerne ikke helt ens. Hvordan de varierer, og hvilken konsekvens det har for analysen, skal du og din makker selv prøve at tænke jer frem til.
Vær omhyggelige med at notere, hvordan I analyserer jer frem til den formodede gerningsperson eller far. Efter analysen af de 3 cases, skal du nemlig besvare en række spørgsmål om, hvem der er gerningspersonen / far, og hvordan du har analyseret dig frem til det.
Hvordan kan man bruge DNA-profiler til at identificere en forbryder? Det skal du prøve at undersøge nu, hvor du skal se, om DNA-spor fra et indbrud kan bruges til at fange en indbrudstyv. Se videoen om indbruddet i Gentofte, og se så om du kan hjælpe politiet med at finde den skyldige.
Inden du går i gang med analysen, skal du overveje følgende: Forventer du, at et evt. DNA-spor er identisk med eller forskelligt fra det DNA-spor, som du får fra den tyv, der har begået indbruddet? Husk at begrunde dit svar.
Nederst på siden finder du en DNA-profil fra et DNA-spor samt DNA-profiler fra 8 mistænkte personer. Analyser DNA-sporet og sammenlign det med det fra de 8 mistænkte. Kan du finde frem til indbrudstyven?
I case 2 skal du prøve at løse en anden forbrydelse, som DNA-profiler kan hjælpe med at opklare, nemlig voldtægt. Udfordringen ved at løse voldtægtsforbrydelser er, at det er vanskeligt at finde DNA-spor i en kvindes skede uden at få celler fra kvindens slimhinde med i prøven. Se videoen og prøv at se om DNA kan identificere, hvem der har misbrugt Naja.
Inden du går i gang med analysen, skal du overveje følgende: Hvad vil det betyde for et DNA-spor, hvis det er blandet med offerets DNA, og hvad skal du gøre for at få et rent DNA-spor fra voldtægtsmanden?
I case 3 skal du prøve at finde faren til to børn. De to børn har to forskellige fædre. DNA-profiler bruges nemlig også til at bestemme, hvem der er den biologiske far til et barn, så faren kan betale børnepenge til moren. Se videoen og find frem til, hvilke to af de tre mulige fædre, der er fædre til Ulrikkes to børn.
Inden du går i gang med analysen, skal du overveje følgende: Hvor får et barn sit DNA fra, og hvad betyder det for et barns DNA-profil?
Her skal du skrive, hvad du har fundet ud af ved at analysere de 3 cases og begrunde, hvorfor analyserne ikke er helt identiske.
- Hvem af de 8 mistænkte i indbrudstyvscasen stammer DaN sporet fra?
- Hvem stammer DNA sporet fra drug rape casen fra?
- Hvorfor kan der være op til 4 chromatogramtoppe for et STR område, når der er tale om voldtægtssager?
- Hvad har du gjort for at identificere hvilke STR alleler som voldtægtsmanden havde?
- Hvorfor er et barns STR alleler ikke identiske med morens STR alleler?
- Hvordan udregnes sandsynligheden for at en person har en bestemt DNA profil?
Modul 4: DNA Phenotyping
I dette modul skal du:
- Lære om, hvorfor vi har forskellige øjenfarver.
- Undersøge, hvor godt Mendelsk nedarvning kan forklare øjenfarve.
- Lære om, hvordan mutationer, der har betydning for øjenfarve, kan identificeres.
DNA phenotyping – eller på dansk DNA fænotypning – er at bruge DNA til at forudsige, hvordan en person ser ud. For politiet giver det mulighed for at få information om en persons udseende ud fra et DNA-spor, som man ikke har kunnet finde et match for i politiets DNA-profil database. Man kan sige, at det svarer til politiets fantomtegninger, men i stedet for en tegning af personen ud fra vidneudsagn, laves tegningen ud fra DNA-sporets information.
Vores udseende er i høj grad bestemt af proteiner. Nogle proteiner fungerer direkte som farvestoffer i vores hud, hår og øjne mens andre er med til at styre hvilke gener, der tændes og slukkes for. Under fostertilstanden og ungdomsårene er det med til at styre, hvordan kroppen udvikles, og hvordan vi ender med at se ud.
I dette modul skal du prøve at finde ud af, hvad der bestemmer en persons øjenfarve, og hvordan man kan finde de steder i en persons DNA, der har mest betydning for en persons øjenfarve. Det lyder måske simpelt, men forklaringen er desværre ikke så nem, som du lærte i folkeskolen.
Du skal nu lære om, hvad der giver en øjenfarve. Se animationen og lær om melanins betydning for øjenfarve.

Du har nu prøvet at matche DNA-spor op mod DNA-profiler og har forhåbentlig hjulpet politiet med at løse to forbrydelser og retten med at afgøre en faderskabssag. Desværre er det langtfra altid, at gerningsmandens DNA ligger i politiets eller den internationale database, eller at det lykkes for politiet at få DNA fra mistænkte. Lad os se på, hvad politiet har af andre muligheder.
Hvis et vidne har set gerningsmanden m/k, vil man ofte bede vidnet om at forklare, hvordan personen så ud og ud fra det tegne en fantomtegning. Ud fra beskrivelsen kan politiet så efterlyse en person med nogenlunde det udseende. Problemet er, at det er meget svært at huske en person, som man har set i kort tid.
Lille parøvelse: I har hver især et halvt minut til at beskrive en lærer på skolen. Personen må kun beskrives ud fra udseende, ikke opførsel. (Det er sjovest, hvis du vælger en lærer, der ikke skiller sig helt ekstremt ud – farvet hår eller hanekam gør denne øvelse for nem).
Lav opsamling i plenum – hvor godt gik det med at identificere lærerne?
Er sporet derfor koldt? Ikke nødvendigvis – for DNA indeholder jo koden for, hvordan vi ser ud.
Diskuter i par: Hvilke træk ville være mest værdifulde for politiet, hvis de skal kunne identificere en forbryder? Hvilke af disse karaktertræk tror I, man kan finde vha. DNA? Skriv jeres vigtigste overvejelser ned i diskussionsfeltet nedenfor.
Et af de fysiske karaktertræk, der vil kunne hjælpe politiet med at identificere en person, er øjenfarve. Øjenfarve bruges tit som et eksempel på Mendels 1. og 2. gens nedarvning i lærebøger. Men hvor godt beskriver den type nedarvning egentlig fordelingen af øjenfarver? Lad os undersøge det ved at se på, hvordan fordelingen af brune, grønne og blå øjenfarver vil være, hvis de følger Mendels 2-gens nedarvning.
Traditionelt beskrives øjenfarve som værende styret af B- og G-genet. Den dominant allel af B-genet giver brune øjne, og den recessive allel b giver blå øjenfarve. For G-genet gælder, at den dominante allel G giver grøn øjenfarve, og den recessive allel g giver blå øjenfarve.
B-genet er dominant i forhold til G-genet. Så hvis der er en dominant B-allel, vil øjenfarven altid blive brun, men er der ikke en dominant B-allel, vil det være G-genet, der bestemmer øjenfarven. Fordi B-genet dominerer over G-genet, kaldes denne type for nedarvning epistasi.

Øvelse:
Du skal nu prøve at se på sandsynligheden for, at et par får et barn med blå øjne. Kvinden har genotypen BbGg og manden har genotypen bbGg. Svar først på spørgsmålene nedenfor.
- Hvilken øjenfarve har kvinden? Hvorfor?
- Hvilken øjenfarve har manden? Hvorfor?
- Hvilke 4 forskellige kombinationer af B og G genet kan der være i kvindens ægceller?
- Hvilke 2 forskellige kombinationer af B og G genet kan der være i mandens sædceller?
Download nu regnearket og lav krydsningsskemaet for nedarvningen. Brug evt. din lærebog som hjælp.
Hvad er sandsynligheden for, at parret får et barn med brune øjne, et barn med grønne øjne og et barn med blå øjne?
Krydsningsskema Mendel (xlxs)
Krydsningsskema Mendel (csv)
Du har nu vist, at sandsynligheden for at få et barn med blå øjne er meget lille, hvis ellers modellen med B- og G-genet er sand. Du skal nu prøve at se på et kort, der viser den procentvise fordeling af blå øjenfarve i Europa.

Er der noget, du undrer dig over?
I eksemplet med Mendel 2. gens nedarvning brugte vi 2 “gener”, B og G-genet, til at forklare nedarvningen. I virkeligheden findes de 2 gener ikke, men hvilke virkelige gener bestemmer øjenfarven, og hvordan finder man dem?
Pathway analysis er en af de metoder, som bruges til at identificere gener og er interessant at undersøge. Se videoen og lær om principperne i, hvordan en Pathway analysis laves.
Ved at se på biosyntesevejen for melanin, har du identificeret enzymet tyrosinase som en mulig årsag til nedsat melanin mængde i øjet. Enzymet kodes af Tyr-genet, og du tænker, at mutationer i dette gen vil kunne føre til et defekt enzym og nedsat eller manglende produktion af melanin.
Du har fået lavet DNA-sekvenser for tyrosinase genet fra 6 personer, hvor du også kender deres øjenfarve. Det skal du nu prøve at analysere og se, hvor godt det kan forklare den observerede øjenfarve.

Du skal nu prøve at undersøge det kodende DNA for Tyr-genet hos 6 personer. Brug programmet ‘Ugene’ til at sammenligne to sekvenser af gangen ved at lave en pairwise alignment. Du skal bruge Tyr_ref_DNA og Tyr_ref_Amino som dine reference sekvenser, dvs den, der svarer til den normale DNA- eller aminosyresekvens. Sekvenserne er vedhæftet som FASTA-filer:
Vejledningen for at lave en pairwise alignment kommer her:
Klik på start page fanebladet. Du får nu følgende 4 valgmuligheder:

Klik på open file(s) ikonet og marker følgende to filer: Tyr_ref_DNA.fa og Mut_1_DNA.fa . Tryk
på open. Du får nu følgende muligheder.

Klik på join sequences into alignment – tryk ok. Du vil nu se følgende skærm.

Vælg fanebladet ‘Actions’ og derefter ‘Align’ og ‘Align with MUSCLE’. Der kommer et nyt vindue op. Lad indstillingerne være og klik på ‘Align’ knappen nederst til højre. De to sekvenser bliver nu placeret, så homologe basepar står under hinanden.

Øverst over sekvenserne er der søjler, der indikerer, hvor ens DNA-baserne er. Da der kun er 2 sekvenser, der sammenlignes, er søjlen enten 100% eller 0%. DNA-baserene er enten ens eller forskellige på positionen. Scroll hen over sekvenserne indtil du via søjlerne kan se, at der ikke er en identisk DNA-base på en position. Noter basepositionen og beskriv, hvilken type mutation, der er sket. Brug figuren øverst som hjælp.
Gentag øvelsen, men denne gang med aminosyresekvenserne. Du bruger Tyr_ref_Aminosyre.fa som reference sekvens og begynder ved at sammenligne med Mut_1_Amino.fa sekvensen. Hvilken betydning får DNA-mutationen i forhold til aminosyresekvensen? Noter effekten.
Gentag for de øvrige 4 mutationer og beskriv også, hvilken type mutation det er, og hvilken effekt de har på aminosyrerækkefølgen. Noter igen effekten ned for alle mutationerne.
I en undersøgelse har man set på 6 personer, hvor person 1 er homozygot dominant for Tyr reference sekvensen, mens de øvrige personer er homozygot recessive for de 5 mutationer, du lige har undersøgt. Øjenfarven for de 6 personer er:
Tyr reference sekvens: Brun
Mutation 1 : Blå
Mutation 2: Brun
Mutation 3: Blå
Mutation 4: Grøn
Mutation 5: Brun
Sammenlign de 5 mutationer med øjenfarverne. Kan forskellene i øjenfarve forklares ud fra effekten, de forskellige mutationer har på Tyr-genet, og derfor også på det færdige tyrosinase enzym?
En anden måde at undersøge, hvilke gener, der spiller en rolle for øjenfarve, er ved at lave et såkaldt Genome Wide Association study. Se videoen og lær mere om, hvad et Genome Wide Association Study kan bruges til.
Du har nu set, at mutationer i OCA2-genet og Herc2-genet har meget stor betydning for øjenfarve, men hvorfor egentlig? I denne lille øvelse skal du læse en kort engelsk tekst og lære om, hvad der vides og ikke vides om OCA2-genets funktion. Herc2-genets funktion kommer du først til at lære om i modulet om aldersbestemmelse, så her må du væbne dig med tålmodighed.
Selve øvelsen: Du skal skrive et kort resume på dansk på ca. 10-15 linjer om funktionen af det protein, som OCA2-genet koder for – P-protein. Inddrag figuren over den engelske tekst i din beskrivelse. Teksten afleveres via submit funktionen nederst på siden.

OCA2 melanosomal transmembrane protein – Normal function
The OCA2 gene (formerly called the P gene) provides instructions for making a protein called the P protein. This protein is located in melanocytes, which are specialized cells that produce a pigment called melanin. Melanin is the substance that gives skin, hair, and eyes their color. Melanin is also found in the light-sensitive tissue at the back of the eye (the retina), where it plays a role in normal vision.
Although the exact function of the P protein is unknown, it is essential for normal pigmentation and is likely involved in the production of melanin. Within melanocytes, the P protein may transport molecules into and out of structures called melanosomes (where melanin is produced). Researchers believe that this protein may also help regulate the relative acidity (pH) of melanosomes, by controlling the flow of chloride ions in and out of the melanosomes. Tight control of pH is necessary for most biological processes, as altered pH may influence the 3D structure of proteins and enzymes within cells and inhibit their normal functions.
Du har nu lært om, hvordan gener for en egenskab som øjenfarve kan findes i genomet. Men hvorfor bruges den samme metode ikke bare altid? Det skal du her til slut prøve at vurdere ved at svare på følgende spørgsmål:
- Hvilke fordele er der ved at bruge pathway analysis, når man skal finde gener, der har med en egenskab at gøre?
- Hvilke problemer er der med metoden?
- Hvilke fordele er der ved at lave et Genome Wide Association study?
- Overvej, hvorfor en metode som Genome Wide Association study ikke altid bruges, selvom den er bedre til at finde de gener, der har en sammenhæng med en egenskab som f.eks øjenfarve.
Modul 5: Forudsigelse af øjenfarve
I dette modul skal du:
- Lære om, hvordan data kan analyseres vha. machine learning.
- Træne et computerprogram til at analysere, hvilke mutationer, der er vigtige for at bestemme øjenfarve.
- Hjælpe politiet med
Du skal i dette modul lave en Machine Learning analyse og hjælpe politiet med at finde øjenfarven fra 3 personer, der har efterladt et DNA-spor på et drabsted. Til analysen skal du bruge et program, der skal installeres på din computer, nemlig ‘Orange’. På denne side er der en vejledning til, hvordan programmet downloades og installeres samt en kort introduktion til, hvordan programmet virker.
Installer programmet ‘Orange’
Programmet Orange hentes her
Installer ‘Orange’ på din computer.
På mac
- Download programmet.
- Åben dmg-filen i din overførselsmappe.
- Træk Orange-ikonet til din Applications mappe.

På Windows
- Download programmet.
- Åbn Orange Setup-programmet og følg instruktionerne – check nedenstående billeder for at se, hvad du skal vælge.
- Vælg “Install for anyone using this computer”.

- Accepter “Installation af required packages” – også Anaconda.

- Tryk “Next” ved de næste step af installationen.
- Afslut Setup-programmet.

Se videoen og hør om, hvad der skete med den kendte pusher og bandekriminelle Benny Albrechtsen.
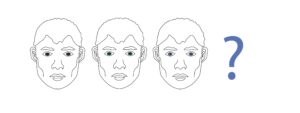
Du skal nu hjælpe politiets retsgenetikere med at lave DNA-phenotyping af de 3 DNA-spor fra Benny Albrechtsens lejlighed. Du har fundet ud af, hvilke gener, der har betydning for øjenfarve i sidste modul, men hvordan vurderer du, hvilken øjenfarve gerningsmændene har, når der er mange forskellige gener, der har betydning for øjenfarve? Og hvor sikker kan du være på, at din forudsigelse er rigtig?
For at løse det problem får du hjælp af politiets IT-eksperter. De foreslår, at du lader computeren gøre det hårde arbejde med at finde ud af, hvilke gener der har mest betydning for øjenfarven. Først skal du fodre computeren med data, så den lærer sammenhængen mellem, hvilke mutationer der giver hvilken øjenfarve. Metoden kaldes Machine Learning, som du skal se en lille video om på næste side.
Se videoen her om Machine Learning og svar på følgende 3 spørgsmål:
- Hvad er forskellen på et almindeligt computerprogram og Machine Learning?
- Hvorfor minder lineær regression om Machine Learning?
- Hvorfor er træningsdata vigtige, når du skal lave Machine Learning?
Det første skridt i din Machine Learning analyse er at importere træningsdata til programmet. Træningsdatasættet for øjenfarvebestemmelse består af 200 personer, hvis øjenfarve er kendt. For hver person er genotypen for de 24 SNP-positioner, der viste stærkest association i Genome Wide Association Study, bestemt.
Data ser ud som vist på tabellen nedenfor: Hver person udgør en række. I første kolonne (label) ses personens øjenfarve. Øjenfarven kan være brun, blå eller andet (grøn eller lysebrun). De næste 24 kolonner viser hver en SNP-position, der er navngivet med rs efterfulgt af 8 tal. For hver person angives om SNP-mutationen findes i ingen (0), den ene (1) eller begge alleler (2).

Du skal nu importere datasættet til din analyse.
Download først filen “Træningsdata_øjenfarve.csv” til din computer.
Træningsdata_øjenfarve (csv)
Åben programmet Orange.
- Tilføj “CSV File Import” fra paletten “Data”.
- Dobbelt klik “CSV File Import” og vælg filen “Træningsdata_øjenfarve.csv”. NB: Filen får måske et andet navn, når du downloader den.
- Vælg “Semicolon” ud for “Cell delimiter”

- Marker alle kolonner og vælg “Categorical” ud for “Column type”

- Klik “Ok”
- Du kan altid komme tilbage til dette vindue ved at klikke på “Import Options…” i “CVS File Import“-vinduet.
- Husk at gemme dit workflow så du ikke mister, hvad du har lavet. Det gøres under fanebladet ‘file’ og derefter ‘save as’.
Du er nu klar til at analysere data og bestemme øjenfarven af DNA-sporene ved hjælp af Machine Learning.
Inden vi træner datasættet, skal du prøve at undersøge de 24 SNP-mutationer for at se, hvor stærk sammenhængen er mellem en genotype og en bestemt øjenfarve.
Tilføj Distributions fra paletten Visualize i ‘Orange’.
- Forbind “CSV File Import” og “Distributions”. Det gøres ved at klikke på den grå halvcirkel til højre for ‘CSV file import’ og så trække en linje hen til den grå halvcirkel til venstre for ‘Distributions’.

Du er nu parat til at se fordelingsstatistik for de 24 mutationer.
- Dobbelt klik på ‘Distributions’.
- I Distributions-vinduet kan du undersøge fordelingen af muterede allleler for de 24 forskellige SNP’er. Sørg for at ‘Split By’ står på none som vist nedenfor. Her kan du se fordelingen for mutationen rs1667394. Der er flere end 140 personer, som har mutationen på begge kopier af genet og lidt færre end 10 personer, som ingen mutationer har på dette gen.

Undersøg de forskellige mutationer på listen. Er der nogen af mutationerne, hvor der er meget få muterede alleler?
Overvej, hvorfor det er vigtigt at se på fordelingen af alleler for en SNP-position, før du bestemmer, om den skal inkluderes i analysen.
Det, som du i virkeligheden er interesseret i, er, om der er en sammenhæng mellem øjenfarve og SNP genotyper. Det kan du undersøge på følgende måde:
Nederst i Distributions-vinduet sættes Split by til label og der sættes flueben i Show probabilities. 
Du ser nu følgende:

Diagrammet viser nu sandsynligheden for de forskellige øjenfarver for hver genotype. Husk, at 0=homozygot for ikke muteret allel af SNP, 1=heterozygot og 2=homozygot for muteret allel af SNP.
For SNP-mutation rs1800414 ses, at fordelingen af brune (rød søjle), andre (grøn søjle) og blå (blå søjle) øjenfarver nogenlunde er ligeligt fordelt mellem de tre genotyper. For denne SNP-mutation er sammenhængen mellem øjenfarve og genotyper derfor ikke særlig stærk.
Se nu alle de andre 23 SNP-mutationer igennem og noter de 3 SNP-mutationer, som ser ud til at betyde mest for øjenfarven.
Når du bruger Machine Learning, skal du vælge en model, som du træner vha. dine træningsdata. Siden øjenfarve typisk opdeles i brun, blå og andet (grøn og lysebrun), er data diskrete, hvilket vil sige, at de inddeles i kategorier. En person har derfor enten brune øjne, blå øjne eller andet. Der findes ikke andre muligheder.
En Machine Learning model, der er meget anvendelig til analyse af denne type data, er Decision Tree modellen. Denne model bruges især til diskrete data, dvs. data, der kan deles op i kategorier såsom brun, blå og andet. Det var derfor, at du markerede data som ‘categorical’, da du importerede datasættet, da det viser, at det er diskrete data.
Træ-modeller deler træningsdatasættet i mindre og mindre grupper, indtil alle eller næsten alle i gruppen har samme label. Her deler vi ud fra SNP-mutationerne, indtil alle i en gruppe har samme øjenfarve.
For hver mutation udregner træ-modellen, hvor stor en procentdel, der har brun, blå og anden øjenfarve. For rs12913832 kan du i tabellen se, at både 0 og 1 mutation stort set kun forekommer for brun og anden øjenfarve. Godt halvdelen med 2 mutationer har blå øjne.
Træ-modellen deler derfor personerne i 2 grupper: Dem med 0 og 1 mutation (mest brune og andet) og dem med 2 mutationer (fleste med blå). Derefter undersøges de 2 grupper hver for sig, og grupperne deles i mindre grupper vha. mutationer for andre gener.
| Øjenfarve | |||
|---|---|---|---|
| Antal mutationer for rs12913832 | Blå | Brune | Andet |
| 0 | 0% | 100% | 0% |
| 1 | 7,8% | 73,4% | 18,8 |
| 2 | 52,1% | 19,3% | 28,6% |
Illustration af Decision Tree
Træ-modellen vises som et omvendt træ, hvor man begynder øverst. I den øverste boks vises, at 43,5% af de 200 personer i datasættet har brune øjne. Derefter deles datasættet ud fra mutationen rs12913832. Den ene gruppe består af 81 personer med 0 eller 1 mutation på rs12913832. Af dem har 79% brune øjne. Af de 119 med 2 mutationer har 52% blå øjne.
Når man bruger modellen til forudsigelser, følger man træet fra top til bund. Hvis vi begynder med en person med 1 mutation på rs12913832, går vi til højre i træet. Da der står “brown” på højre boks, vil vi forudse, at personen med 79% sikkerhed har brune øjne.

Du skal nu træne en Machine Learning model, så du kan forudsige øjenfarve ud fra dit træningsdatasæt.
Du skal nu træne ML-programmet med dine træningsdata og med Decision Tree som model.
Åben dit workflow fra statistik over mutationer.
- Tilføj ‘Select Columns’ fra paletten ‘Transform’.
- Forbind ‘CSV File Import’ og ‘Select Columns‘.

- Dobbelt klik på ‘Select Columns’.
- I ‘Select Columns’ vinduet flyttes ‘label‘ til Target-feltet. Dermed har du valgt, at modellen skal trænes til at forudse øjenfarve – luk vinduet.

- Tilføj ‘Tree’ fra paletten ‘Model’.
- Forbind ‘Select Columns’ og ‘Tree’.
- Vælg ‘Predictions’ fra paletten ‘Evaluate’.
- Forbind både ‘Select Columns’ og ‘Tree’ til ‘Predictions’.
Dit workflow skal se sådan ud:

Datasættet er nu blevet brugt til at lave en Machine Learning model. Men inden vi kan bruge modellen til at forudsige øjenfarven ud fra DNA-sporene, skal du lige tjekke om modellen faktisk også er god til at forudsige øjenfarven.
- Dobbeltklik på ‘Predictions’.
- Vælg ‘Classes in data’ ud for ‘Show probabilities for’. Så får du modellens præcision for forudsigelserne som sandsynligheden for de forskellige øjenfarver.
- Gør ‘Tree’ kolonnen bredere så det ser ud som vist nedenfor.
Under ‘Label’ står de 200 testpersoners øjenfarve. Under ‘Tree’ står modellens forudsigelse. Tallene i den første række viser, at modellen angiver 0% for blå, 96% for brun og 4% for anden øjenfarve.
- Se på de første 20 personer i testdatasættet. Hvor mange af dem kan ML-modellen korrekt forudsige øjenfarven på?

Du er nu klar til at analysere DNA-spor, hvor øjenfarven ikke er kendt.
Du har nu trænet din ML-model og er klar til at hjælpe politiet med ny viden om gerningsmændene fra mordet på Benny Albrechtsen. Ved hjælp af PCR- og DNA-sekventering har du fået bestemt genotypen for de 23 mutationer, som du bruger i din model fra de tre DNA-spor fundet på gerningsstedet. Resultatet ligger i csv-filen i det følgende.
Fortsæt i det workflow du har lavet i ‘Orange’ på sidste side (Træning af ML model).
- Tilføj ‘CSV File Import’ fra paletten ‘Data’.
- Dobbelt klik på ‘CSV File Import’ og indlæs DNA-sporene i filen ‘DNA_spor_farve.csv’. NB: filen får måske et andet navn, når du downloader den.
- Husk at vælge ‘Semicolon’ i ‘Cell delimiter’ og ‘Categorial’ i ‘Column Type’.
- Tilføj ‘Select Columns’ fra paletten ‘Transform’.
- Forbind ‘CSV File Import’ og ‘Select Columns’.
- Dobbelt klik på ‘Select Columns’.
- Check at alle 23 mutationer er placeret under “Features”
Nu er du klar til at bestemme øjenfarven.
- Tilføj ‘Predictions’ fra paletten ‘Evaluate’.
- Forbind ‘Select Columns‘ til ‘Predictions‘.
- Forbind ‘Tree’ fra den tidligere opgave til den nye ‘Predictions’.
Dit workflow skal se sådan ud:

- Dobbelt klik på ‘Predictions’.
- I ‘Predictions’ vinduet skal du vælge ‘Classes known to the model’

og gøre Tree-kolonnen bredere så du kan se sandsynlighederne. - Noter, hvilken øjenfarve modellen forudser for DNA-spor 1, 2 og 3. Du skal bruge denne viden senere.
- Hvilke sandsynligheder angives for de 3 mulige øjenfarver for DNA-spor 1?
Når du har noteret alle øjenfarver og deres sandsynligheder, skal svarene sendes ind til politiet. Det gør du ved at svare på spørgsmålene nederst på siden.
Modul 6: Aldersbestemmelse
I dette modul skal du:
- Lære om, hvad epigenetik og genekspression er.
- Analysere DNA for DNA-methyleringer.
- Lære, hvordan variation i DNA-methyleringer kan bruges til aldersbestemmelse.
- Bestemme alderen for de 3 DNA-spor fra Benny Albrechtsens lejlighed.

Selvom politiet ikke kan matche et DNA-spor i deres DNA profildatabase, har du indtil videre set, at politiet alligevel kan finde information om en persons udseende såsom øjenfarve. Men faktisk sladrer DNA også om en persons alder. Det skal du lære om i denne øvelse.
Nogle proteiner dannes i forskellige mængder på forskellige tidspunkter i en persons liv. Det betyder, at der skrues op eller ned for proteinsyntesen af disse proteiner afhængigt af, hvor gammel du er. Mængden af protein, der dannes på et bestemt tidspunkt, styres af, hvor meget transkription der sker af genet, der koder for proteinet. En proces, der samlet kaldes for genekspression.
En måde genekspression styres på er ved kemisk at blokere for bestemte baser i DNA’et. Når DNA baserne blokeres, forhindres den normale funktion af DNA-området, der både kan være at fremme eller hæmme genekspression. Disse ændringer kaldes epigenetiske, da der ikke sker DNA-mutationer, men kun blokering af nogle af DNA-baserne, hvilket skyldes miljøpåvirkninger.
I denne animation skal du lære om, hvordan genekspression foregår. Efter at have set videoen skal du svare på følgende 3 spørgsmål:
- Hvad er et enhancer område?
- Hvorfor kan mutationer i ikke kodende introns alligevel påvirke genekspressionen af andre gener?
- Hvorfor medfører mutationen i HERC2 intronet, at en person får blå øjne?
Hos mennesker er det hyppigst cytosin, der blokeres ved, at der sættes en methylgruppe på basen ved hjælp af enzymet DNA methyltransferase. Det mest almindelige er, at methylgruppen sættes på position 5 i cytosin, som det kan ses på figuren. Methyleringer er ikke permanente, da methylgruppen også kan fjernes igen af enzymet demethylase.

Ved at analysere fordelingen af DNA-baser har forskere opdaget, at der spredt ud over genomet er områder på ca. 200 basepar, der har et højere % indhold af C- og G-baser end andre områder. Disse områder kaldes CpG islands, hvor CpG betyder, at C- og G-baserne kommer lige efter hinanden med en phosphat imellem. CpG islands er meget ofte en del af promoterregioner, hvilket er områder før gener, hvor RNA-polymeraser kan sætte sig, når de skal lave transkriptionsdelen af proteinsyntese.
Alt efter en persons alder vil der være forskelle i antal cytosin baser i CpG island områderne, der vil være methyleret. Om der bliver flere eller færre cytosin methyleringer varierer alt efter, hvilket gen CpG island regionen er forbundet med. Du skal nu se på, hvordan methyleringsgraden kan analyseres, og hvordan det kan bruges til at estimere en persons alder.
n måde at bestemme methyleringsgraden af et DNA-område er ved at behandle DNA-prøven med bisulphit, \(HSO_3^-\). Cytosin, der ikke er methyleret, vil ved behandling med bisulphit omdannes til RNA basen uracil. Methyleret cytosin (5-methylcytosin) omdannes ikke ved behandling med bisulphit. Ved at behandle DNA-sporet med bisulphit vil alle ikke methylerede cytosin baser derfor ændres til uracil.

For at kunne se, hvilke cytosin baser, der ændres til uracil, og hvilke der ikke gør, er det nødvendigt at lave et kontrolforsøg. DNA-sporet deles derfor i to, og der laves PCR-reaktion på både bisulphit behandlet og ikke-bisulphit behandlet DNA. Sammenlignes DNA-sekvenserne fra de to prøver, vil alle de ikke methylerede cytosin baser være ændret fra C til U (pga. PCR opformeringen vil det dog i DNA-sekvensen blive aflæst som et T), mens alle de methylerede cytosin baser ikke vil være ændrede.
Så er det din tur. Se på de to DNA-sekvenser fra en bisulphit behandlet prøve og en kontrolprøve. Hvilke cytosin baser har oprindeligt været methylerede og hvilke har ikke? Tag et screenshot, når du har lavet sammenligningen, og upload dit svar her på siden.

Kontrol DNA:
GGCGCTGGGGTTCCGGCCCT
Bisulphit DNA:
GGCGTTGGGGTTCTGGTCCT
Du skal nu prøve at bestemme alderen af de 3 DNA-spor fra drabet på Benny Albrechtsen ved at lave en analyse af graden af DNA-methylering. Første skridt i analysen er at udregne, hvor mange af cytosinbaserne i DNA-området, der skal analyseres, der er methyleret. Det gøres ved at sammenligne resultatet af den bisulphit behandlede prøve og den almindelige prøve for det DNA-område, der skal undersøges og derpå udregne, hvor stor en procentdel af cytosinbaserne, der er methyleret.
Nedenfor er vedhæftet to filer, der indeholder resultatet af et udsnit af DNA-sekvenseringen af DNA-området CCDC102B for en person. CCDC102B er et af de DNA-områder, der kan bruges til at estimere en persons alder. Begynd med at sammenlige dem ved at downloade dem til din computer og gemme dem i en folder, så du kan finde dem.
Komprimeret arkiv (ZIP fil)
Klik nu på Open File(s) i Ugene.

Vælg den folder, hvor du har placeret de downloadede filer og marker dem begge to.

Klik på “Open”. Du ser nu følgende:

Klik på “Join sequences into alignment” og klik “OK”. Den ubehandlede og behandlede DNA-sekvens placeres nu under hinanden, så du kan sammenligne baserne i alle positionerne i DNA-sekvensen.

På figuren overfor er vist de 17 basepar af CCDC102B DNA-sekvenserne for den ubehandlede og bisulphit behandlede sekvens. Alle basepar, som er ens, markeres med en fuld søjle, men på position 14 er C’et i den ubehandlede prøve omdannet til et T (U). Dette C-nucleotid har derfor oprindeligt ikke været methyleret.
Scroll hen over din alignment og noter, hvor mange C-nucelotid positioner, der ikke har været methyleret i CCDC102B DNA-sekvensen. Svaret skal skrives ind i quizzen nederst.
Du skal nu udregne, hvor mange procent af cytosin baserne, der er methyleret. Åben filen CCDC102B DNA ubehandlet.fa. i Ugene via “Open file(s). Du får nu vist DNA-sekvensen for DNA-området CCDC102B. Ser du ikke sekvensen trykkes på det hvid/grønne ikon med ACG i øvre højre hjørne. For at analysere sekvensens indhold af cytosin nucleotider trykkes på søjlediagram ikonet yderst til højre. En statistikboks åbner sig med 5 valgmuligheder. Åben den næstøverste, “Character Occurence”, ved at klikke på den.

Noter CCDC102B DNA-sekvensens antal af C-nucleotider (faktiske antal, ikke procenttal).
Åben nu filen CCDC102B DNA bisulphit behandlet.fa. i Ugene og noter igen antallet af C-nucleotider i DNA-sekvensen.
Du kan nu udregne methyleringsgraden i procent på følgende måde. Da de ikke methylerede cytosin nucleotider omdannes til uracil (i sekvensen vist som thymin T), er antallet af methylerede cytosin nucleotider lig med antallet af C-nucleotider i den bisulphit behandlede prøve. Du udregner den procentvise methyleringsgrad ved at dividere antallet at methylerede C-nucleotider med antallet af C-nucleotider i den ubehandlede prøve og gange resultatet med 100. Noter resultatet/ svar på spørgsmålene:
Hvor mange af cytosin baserne i CCDC102B DNA sekvensen er ikke methyleret?
Hvor stor er methyleringsgraden for CCDC102B DNA sekvensen?
Du har nu regnet dig frem til, hvor stor en del af CCDC102B DNA-området, der er methyleret for en person. Men for at du kan bruge denne viden til at estimere alderen af en person ud fra deres DNA-spor, kræver det, at du kender methyleringsgraden for et stort antal af personer med forskellige aldre. Derfor skal du nu prøve at analysere sammenhængen mellem methyleringsgraden af området CCDC102B i personernes DNA og alder for 100 personer og ud fra det bestemme alderen af et DNA-spor.
- Åben data fra filen “CCDC102B.xlsx” i Excel eller CCDC102B.csv i Google sheets. Filerne er vedhæftet nederst på siden. Du kan også vælge at importere filerne til dit foretrukne matematik program.
- Afbild data for de 100 personer i et X-Y diagram med alderen som den uafhængige variabel (x-værdi) og methyleringsgraden som den afhængige variabel (y-værdi)
- Lav en tendenslinje og få den bedste ligning og \(R^2\) værdien vist. Vurder hvilken af følgende funktioner, der beskriver data bedst: Lineær, eksponentiel eller logaritmisk. Vurder det ved at sammenligne \(R^2\) værdien for de tre funktioner.
- I en DNA-prøve bestemmes methyleringsgraden for CCDC102B-området til 22,5. Beregn alderen for denne person vha. den regressionsmodel, der beskrev data bedst. Brug den bestemte ligning til at udregne alderen.
- En person er 20 år. Bestem vha. modellen methyleringsgraden for CCDC102B området svarende til denne alder.
- Hvor godt vil du mene, at modellen kan estimere DNA sporets alder? Begrund dit svar ud fra analysen af data, som du har lavet.
Som du har set ved regressionsanalysen af DNA methyleringsgraden for DNA-området CCDC102B, er bestemmelsen af alder ud fra et DNA-område ikke særlig præcis. Derfor har forskere identificeret en række DNA-områder, der har varierende DNA methylering i forhold til alder, og som i lighed med området CCDC102B udviser en lineær sammenhæng. Du skal nu prøve at optimere alderbestemmelsen ved at inddrage de ekstra data, for at se om det kan forbedre forudsigelsen.
Da du har flere datasæt, der skal analyseres samtidigt, kan du igen med fordel bruge Machine Learning. Som i eksemplet med øjenfarve skal du derfor først træne en Machine Learning model med et datasæt for at gøre programmet i stand til at forudsige alderen ud fra det givne data. Men til forskel fra øjenfarve er data denne gang numeriske, hvilket gør, at du skal bruge en anden model.
For at vurdere betydning af datamængde, skal du først prøve at lave modellen med kun et DNA-område. Efterfølgende skal du øge antallet af DNA-områder, der bruges til modellens træning, for at se hvilken betydning det har for bestemmelsen. Til slut skal du bruge den mest optimale model til at forudsige alderen på de 3 DNA-spor fra mordet på Benny Albrecthsen.
Hent data ind i Orange
- Nederst på denne side finder du et link til filen “Training.csv“. Hent filen ned på din computer.
- Tilføj “CSV File Import” til dit workflow. Du finder ikonet til venstre i paletten “Data”.
- Dobbelt klik på “CSV File Import”. Klik derefter på mappen med de tre prikker.

- Find filen “Training.csv” på din computer.
- Vælg “Semicolon” ud for “Cell delimiter”.
- Sæt “Decimal” til et komma i stedet for et punktum.
- Marker alle kolonner, klik på “Column type” og vælg “Numerical”.

- Data placeres nu i 8 kolonner:
Den første kolonne er alder for personerne. De 7 andre kolonner viser methyleringsgraden for 7 områder af DNA. De 7 områder er valgt, så der er en tydelig sammenhæng mellem alder og methyleringsgrad. - Klik “Ok”.
Vis de importerede data
- Tilføj “Scatter Plot” fra paletten “Visualize”.
- Forbind “CSV File Import” til “Scatter Plot”.

- Dobbelt klik på “Scatter Plot”. Nu kan du se data i et plot og vælge, hvordan du vil undersøge data.

Vælg hvilke variable du vil vise på x- og y-aksen.
– Vælg “Alder” på y-aksen.
– På x-aksen vælges en ad gangen de syv områder af DNA (CCDC102B, COL1A1, MEIS1-AS3, FHL2, IGSF11, PDE4C og ASPA). - Undersøg hvilken af de 7 områder som du tænker vil være bedst til at forudse alder.
Bestem hvilke data, du vil inkludere i ML-modellen
- Tilføj “Select Columns” fra paletten “Transform”

- Forbind “CSV File Import” til “Select Columns”.
- Dobbelt klik på “Select Columns”.
- Check, at de data, som du vil bruge som input til forudsigelserne, står under features.
- Flyt “Alder” til “Target”, siden det er alder, som du med modellen vil forudse.
- Flyt “CCDC102B” til “Features”. I første omgang bruger vi kun 1 område af DNA til ML-modellen.

- Luk vinduet.
Training (CSV)
Valg af Machine Learning model
Til bestemmelsen af øjenfarve brugte du en træ-baseret model. Træ-baserede modeller egner sig godt til at forudse en kategori (brun/blå/andet øjenfarve) ud fra egenskaber, som også er kategorier, her 0, 1 eller 2 mutationer i hvert af DNA-områderne, som indgår i modellen.
I dette modul skal vi forudse en værdi (alder) angivet som et reelt tal. Modellen skal bruge egenskaber, som også angives som reelle tal, her methyleringsgraden. Derfor skal vi bruge en ny type ML-model: Lineær regression.
ML-modellen “Linear Regression”
ML-modellen “Linear Regression” har samme navn, som den lineære regression du kender fra matematik, men som ML-model kan den noget mere. Den kan bl.a. bruge flere egenskaber samtidigt til at forudse alderen.
Her vil vi ikke gå i dybden med, hvordan modellen virker, da det vil være for omfattende. Du kan forestille dig, at det \(x\), som indgår i udtrykket \(y=a \cdot x + b\), består af flere variable på samme måde, som i funktioner af 2-variable \(f(x,y)\) som du måske allerede kender fra undervisningen i Matematik A.
Lav en simpel ML-model med “Linear Regression”
- Tilføj “Linear Regression” fra paletten “Model”.

- Forbind “Select Columns” til “Linear Regression”.
- Dobbelt klik på “Linear Regression” – sæt fluebenet ud for “Fit intercept”.
Dette er vigtigt – ellers låses skæringen med y-aksen til 0
– svarende til \(b\)-konstanten for den rette linje \(y=a \cdot x + b\) sættes til 0.
Hvis fluebenet ikke er sat, virker modellen dårligt.
- De andre parametre bestemmer, hvordan modellens beregninger virker. Vi kan bruge standard indstillingerne, så her skal du ikke ændre noget.
- Luk vinduet.
Forudse alder ud fra DNA-data
- Tilføj “Predictions” fra paletten “Evaluate” til dit workflow.
- Forbind “Select Columns” til “Predictions”.
- Forbind også “Linear Regression” til “Predictions“.
- Dobbelt klik på “Predictions”.
- Undersøg, hvilken alder ML-modellen forudser, når vi kun har inkluderet data fra området “CCDC102B”

Forudsigelsen af alderen finder du i kolonnen “Linear Regression”. Den faktiske alder for personen finder du i kolonnen “Alder”. - Hvor store afvigelser kan du finde mellem den faktiske alder og den forudsete alder?
Vis resultatet af den simple ML-model
- Tilføj et “Scatter Plot” til dit workflow
- Forbind “Predictions” til “Scatter Plot”
- Nu skal dit workflow se ud som på figuren

- Dobbelt klik på det nye “Scatter Plot” – vis den faktiske “Alder” på x-aksen og modellens forudsigelse “Linear Regression” på y-aksen.

- Sæt flueben ved “Show regression line”

Den lille label på regressionslinjen viser korrelationskoefficienten r for grafen. Hvis der er en perfekt lineær sammenhæng mellem faktisk alder og forudset alder, er \(r=1\). Hvis der ingen sammenhæng er vil vi få \(r=0\). NB: her er \(r\) ikke forklaringsgraden \(r^2\), som du kender fra matematik.
Svar på de 3 forståelses spørgsmål nederst.
- Beskriv grafen for din model.
- Hvilken hældningen skal grafen have, hvis modellen skal være god til at forudse alder ud fra DNA-data?
- Hvordan skal punkterne være placeret i forhold til regressionslinjen, hvis modellen skal være præcis?
Brug af flere DNA-områder i ML-modellen
Indtil videre har vi kun brugt methyleringsgraden i DNA-området CCDC102B til træning af ML-modellen. Nu vil vi forbedre modellen ved at træne ML-modellen med flere af de 7 DNA-områder, vi har data fra.
Opgave – forbedring af ML-modellen
Brug det workflow du allerede har lavet.
- Dobbelt klik på “Select Columns”
- Tilføj et DNA-område “COL1A1” til listen med egenskaber (Features), som modellen bruger til at forudse alderen. Luk vinduet igen.

- Dobbelt klik på “Scatter Plot” og undersøg om modellen er blevet bedre ved at tilføje et ekstra DNA-område til modellen. Hvordan har korrelationskoefficienten og hældningen af regressionslinjen ændret sig?
- Tilføj løbende flere DNA-områder til modellen og undersøg, om modellen bliver bedre til at forudse alderen.
Spørgsmål:
- Virker modellen bedst, hvis du inkluderer alle DNA-områder i modellen?
- Er der et eller flere områder, du bør udelade i modellen?

Du har nu optimeret modellen og er igen klar til at hjælpe politiet med de 3 DNA-spor fra drabsagen på Benny Albrechtsen. Methyleringsdata fra de tre DNA-spor finder du nederst på siden. Gør nu følgende:
- Tilføj en ny “CSV File Import” fra paletten “Data”
- Hent filen “DNA-spor aldersbestemmelse.csv” ned på din computer
- Dobbelt klik på den nye “CSV File Import” og indlæs det nye datasæt ved at vælge filen “DNA-spor aldersbestemmelse.csv”
- Tilføj en ny “Predictions” fra paletten “Evaluate”
- Forbind “Linear Regression” til den nye “Predictions“
- Forbind den nye “CSV File Import” til den nye “Predictions“
Dit workflow skal ende med at se sådan her ud:

Dobbeltklik nu på den nye predictions. Du kan nu aflæse den estimerede alder af de 3 DNA-spor. Noter igen alderen på gerningsmændene fra DNA spor 1, DNA spor 2 og DNA spor 3 ned. Denne viden skal du bruge senere.
Overvej følgende 3 spørgsmål:
- Hvor stor en hjælp vil det være for politiet, at de kender til en gerningsmands alder?
- Hvad mangler politiet at få at vide, før at de kan bruge din aldersestimering til noget?
- Graden af DNA methylering kan variere i forskellige væv i kroppen. Hvilken betydning vil det have for aldersestimeringen af et DNA-spor?
Modul 7: Slægtskabsanalyser
- Lære om, hvordan DNA SNP array microchip bruges til at kortlægge SNP variation hos mennesker.
- Analysere, hvordan DNA nedarves, og hvor meget DNA du deler med din familie.
- Forstå meioses betydning for DNA lighed og forskelle i slægtninges kromosomer.
- Lære om, hvordan SNP variation kan bruges til at udlede slægtskab mellem familiemedlemmer.
- Bruge al den information du har fundet om de tre DNA-spor fra Benny Albrechtsens lejlighed til at identificere gerningsmændene fra forbrydelsen.
Du har nu set, at biologisk køn, fysiske egenskaber som øjenfarve og en persons alder kan estimeres ud fra et DNA-spor. Men der er faktisk en sidste mulighed for at identificere en person, hvis der ikke er et match i en DNA-database. Det er at lave DNA slægtskabsanalyser og bruge dem til at identificere en mistænkt.
I dag er der et stort marked globalt for genealogi DNA-tests. Genealogi er udledning af slægtskab, hvilket traditionelt undersøges vha. fødsels-, døds- og ægteskabsattester. Men siden år 2000 har det været muligt for privatpersoner at sende et mundskrab af slimhindeceller ind til firmaer, der kortlægger ca. 800.000 af de mest almindelige SNP’er hos personen. Det kan i dag gøres for under 500 kr.
Informationen i en sådan analyse kan bruges til at udlede slægtsskab, men også til at identificere familien til en mistænkt. Du skal i dette modul lære om, hvordan slægtsskabsanalyser laves, og hvordan de kan bruges til at identificere forbrydere. Til slut skal du se, om du kan bruge din viden fra DNA-phenotyping af øjenfarve, aldersbestemmelse og slægtsskabsanalyse til at identificere gerningsmændene til mordet på Benny Albrechtsen og hjælpe med at få dem stillet til ansvar for deres gerning.
Første led i en slægtsskabsanalyse er at kortlægge mere end 700.000 SNP positioner fra DNA-prøven fra prøvepersonen. DNA’et skal dog først forbehandles, inden det kan analyseres. Derfor tager en DNA microchip SNP array analyse typisk tre dage at lave, hvor de to første dage går med at forberede DNA’et til selve analysen.
Det genomiske DNA oprenses først, hvorefter det mangedobles ved en metode, der hedder “Whole Genome Amplification”. Siden det er hele genomet, der opformeres, så er det ikke PCR-metoden, der bruges, men resultatet svarer til en PCR-reaktion, men bare for hele genomet. Ligesom for PCR-reaktionen er formålet at sikre, at der er nok DNA til at lave analysen.
Når DNA’et er opformeret, skæres det i mange små stykker vha. en blanding af restriktionsenzymer eller ved ultralyd. De dannede DNA fragmenter denatureres, så de ender med at være enkeltstrengede. DNA prøven er nu klar til at blive analyseret på DNA microchippen. Dette skal du se nærmere på ved hjælp af en animation i selve modulet.

- Oprenset DNA.
- Opformeret genomisk DNA.
- DNA fragmenter efter skæring med restriktionsenzymer eller ultralyd.
- Denatureret enkeltstrenget DNA.
Hvordan virker en DNA microchip? Hvor mange SNP’er kan den analysere samtidigt? Og hvordan kan der skelnes mellem, om en person er homozygot med en allel, homozygot med den anden allel eller heterozygot for en SNP? Dette skal du lære mere om ved at se den ovenstående video.
Du skal nu se, om du har forstået, hvordan en DNA-mikrochip bruges til SNP genom analyser. Svar på spørgsmålene for at få lov til at gå videre i øvelsen.
Hvor mange SNP positioner i genomet kan en standard DNA microchip analysere Per person?
- Ca. 300.000
- Ca. 500.000
- Ca. 700.000
- Ca. 1.000.000
De enkeltstrengede DNA fragmenter på en silica kugle på microchippen binder DNA fragmenter fra hvor mange SNP’er?
- 1
- 2
- 3
- >50
DNA fragmenter på silicakuglen er komplementære til hvilke DNA baser i genomet?
- 50 baser lige før SNP’en
- 50 baser lige efter SNP’en
- Der ligger 25 baser før og 25 baser efter SNP’en
Hvad er DNA polymerasens funktion i forhold til en DNA microchip?
- At lave DNA fragmenter på silicakuglerne
- At sætte en fluorescerende DNA base på DNA fragmenterne på silicakuglen
- Af opformere det genomiske DNA fra DNA prøven, så det kan analyseres
Hvilken farve vil en heterozygot AT SNP lyse op med?
- Grøn
- Blå
- Gul
- Rød
For at kunne studere slægtskab afbildes familie relationer i nedarvningsskemaer. Du har læst om nedarvningsskemaer i lektien til i dag. Se nu på følgende nedarvningsskema og svar på spørgsmålene nederst på siden.

- Hvad viser en cirkel? (en kvinde/en mand)
- Hvad betyder en vandret streg, der forbinder to personer? (at de er søskende/ at de er et par)
- Hvor mange børn har II-1 og II-2 fået sammen? (3/4/5)
- Hvad betyder det, at en person er markeret halvt farvet, halvt hvid? (at personen er syg/ at personen er rask, men bærer en sygdomsallel/ at personen er rask)
- Hvad er kønnet på den person, der er syg? (kvinde/mand)
- Hvor stor er risikoen for at II-5 og II-6 får et sygt barn, hvis de får endnu et barn sammen? (25% /50% /75% /100%)
Hvor meget DNA har du egentlig til fælles med din mor og far? Og dine bedsteforældre? Det skal du lære mere om i denne video.
OBS: Læs teksten nedenunder før I ser animationen.
Selvom du har fået 50% af dit DNA fra din mor og 50% af dit DNA fra din far, er dine kromosomer ikke identiske med din fars ellers din mors. Det skyldes måden kønsceller dannes på. I denne øvelse skal du forklare de forskellige trin i meiosen (kønscelledannelsen) for at forstå, hvorfor dine kromosomer er anderledes end dine forældres.
I skal nu se en animation af meiosen. For at gøre det mere overskueligt er der i animationen kun vist 1 ud af de 23 par kromosomer i en persons DNA. Ligeledes er det kun kernemembranen, der vises, og ikke de enkelte cellers cellemembran.
Se animationen om, hvordan meiosen foregår og forklar til hinanden, hvad der sker i hver af de følgende faser:
DNA replikation
1. meiotiske deling:
Profase
Metafase
Anafase
Telofase
2. meiotiske deling:
Profase
Metafase
Anafase
Telofase
Spørgsmål: Hvilken del af meiosen er skyld i, at dine forældres kromosomer og dine ikke er identiske?
På grund af overkrydsning vil længden af DNA-segmenter, der er identiske på kromosomer blive kortere, jo mere fjernt beslægtet du er med et familiemedlem. Samtidig har du kun fået halvdelen af dit DNA fra hver af dine forældre, hvilket betyder, at du kun har en ud af to alleler for hver position i dit DNA. Så hvordan bestemmes det om et DNA-segment kan stamme fra en slægtning eller ej?
Der findes forskellige metoder til at analysere det, men du skal her lære om den mest simple kaldet Segment approach. Her bruger efterforskere igen muligheden for at sammenligne Single Nucleotide Polymorphisms (SNP’s) på kromosomerne. SNP’erne ligger spredt ud over genomet med en række basepar liggende mellem sig. Hvis 2 personer har den samme allel på en SNP og også på en nabo SNP antages det derfor, at stykket imellem er ens mellem de 2 personer.
Når DNA lighed sammenlignes er der følgende 3 muligheder, når 2 personers DNA sammenlignes (X angiver DNA basepar imellem de 2 SNP’er):

Ens alleler på begge personers SNP: Her er begge personer homozygote for allelen T for SNP 1. Fordi der er ens alleler, vil person 1 og 2 godt kunne have arvet allelen af en eller flere fælles slægtninger.
En ud af to alleler er ens. Her er person 1 heterozygot for henholdsvis SNP 2+3, hvorimod person 2 er homozygot for SNP 2+3. Men da der er en delt allel for SNP 2 (G) og SNP 3 (C) kan det ikke udelukkes, at allelen skyldes en fælles slægtning.
Ingen fælles alleler på en SNP. Her er person 1 homozygot for SNP4 (TT) og begge alleler er forskellige fra person 2 alleler for SNP 4 (AA). Det tolkes som, at der ikke er en fælles slægtning, der har givet denne SNP allel videre.
Se nu på følgende DNA fragmenter fra 2 personer. Brug den beskrevne metode ovenfor og angiv, hvor mange og hvor lange DNA-fragmenter, der er ens. Udelad DNA-fragmenter mindre end 3 nabo SNP’er lange – disse er for usikre, da DNA jo kun er 4 basepar, og derfor er der en hvis sandsynlighed for, at man rent tilfældigt har den samme allel (I virkeligheden udelader man fragmenter noget længere end det). X betyder en række basepar mellem 2 SNP’er.

Du skal svare på 4 spørgsmål ved at analysere 4 resultater, som en yngre kvinde har fået efter at have uploadet data fra en genealogisk undersøgelse til en slægtsskabsdatabase. Resultaterne ses på billedet nedenfor, hvor personernes 23 kromosompar er placeret under hinanden afsluttende med X-kromosomet. Steder, hvor DNA segmenterne er ens er farvet, mens steder, hvor der ikke er DNA-segment lighed, er grå.
Resultaterne stammer fra følgende fire personer: Kvindens far, kvindens bedstemor på morens side, kvindens oldefar på morens side og kvindens grandkusine (kvindes bedstemors søsters barnebarn, på engelsk second cousin).

- Hvilken person er kvindens far? Begrund dit svar.
- Vil kvindens bedstemor, kvindens oldefar eller kvindens grandkusine have færrest DNA segment ligheder med kvinden? Begrund dit svar?
- Hvordan kan du se, at person 2 må være kvindens bedstemor og ikke hendes bedstefar? Begrund dit svar.
- Genealogiske DNA test kan maksimalt spore slægtninge der svarer til dine oldeforældres søskendes oldebørn (på engelsk third cousins). Prøv at forklare hvorfor ud fra figuren med de 4 personers DNA segment ligheder.
På trods af din hjælp med at bestemme de 3 DNA-spors øjenfarve og alder, er det ikke lykkedes for politiet at identificere mulige mistænkte. Du bliver derfor bedt om at lave en slægtsskabsanalyse på de 3 DNA-spor og se, om det giver politiet nogle nye ledetråde i forhold til at opklare drabet på Benny Albrechtsen.
Efter af have lavet en DNA microchip SNP analyse af de 3 DNA-spor prøver du at matche dem mod en slægtskabsdatabase. Du er heldig, for alle tre DNA-spor deler DNA med en person i databasen. Ved hjælp af grundig research får du lavet slægtskabstræer for de tre familier, som har en person, der deler DNA med de tre DNA-spor fra Benny Albrechtsens lejlighed.
Du skal nu prøve at spore de tre gerningsmænd ved at analysere slægtskabs data og kombinere det med den viden, du har om gerningsmændenes øjenfarve og alder. På de næste tre sider kan du se resultaterne af slægtskabsanalysen samt stamtræet for familierne, hvor en af medlemmerne har DNA match med DNA-sporet. Husk, at hver gang du går en generation frem eller tilbage, vil der kun være ca. 50% DNA lighed mellem familiemedlemmer og antag at der er 50% lighed mellem søskende (det vil der ikke altid være i den virkelige verden). Den viden får du brug for, når du skal vurdere, hvem der kan være gerningsmanden.
 DNA spor 1 har DNA lighed med følgende person i DNA slægtskabsdatabasen: Jørgen Karlhøj Mikkelsen. Jørgen er 53 år gammel og arbejder som elektriker i en større produktionsvirksomhed.
DNA spor 1 har DNA lighed med følgende person i DNA slægtskabsdatabasen: Jørgen Karlhøj Mikkelsen. Jørgen er 53 år gammel og arbejder som elektriker i en større produktionsvirksomhed.
Resultatet af analysen ses på kromosomkortet til højre. Identisk DNA er markeret med rødt.
Vurder først graden af DNA lighed. Ser det ud som om der er 50%, 25%, 12,5% eller mindre?
Slægtskabstræet for familien ses nedenfor. Jørgen Karlhøj Mikkelsen er markeret med en blå firkant.

Du ved, at alle 3 DNA-spor stammer fra mænd.
Baseret på din analyse af DNA lighed, hvilke personer i slægtskabstræet kan DNA-spor 1 så stamme fra?
Husk, at du skal tælle personer i en generation fra venstre mod højre – Jørgen Karlhøj Mikkelsen skal derfor skrives som IV-12. De opgivne personer i dine svar skal skrives på samme måde.
Da der er ca. 50% DNA lighed mellem Jørgen Karlhøj Mikkelsens DNA og DNA spor 1, kan DNA’et fra gerningsmanden kun stamme fra generationen før eller efter Jørgen Karlhøj Mikkelsens. Da du ved, at DNA-sporene stammer fra en mand, giver det tre mulige mistænkte: III-9, V-12 og V-15.
Nu skal du bruge din viden om DNA-spor 1. Begynd med at se på dine noter. Hvilken øjenfarve og hvilken alder er blevet estimeret ud fra DNA-spor 1?
Nederst på siden ligger der en samlet PDF med de data, som det er lykkedes politiet at indhente om de tre personer III-9, V-12 og V-15. Læs dem igennem og brug dem til at vurdere, hvem er den mest sandsynlige gerningsmand?
 DNA-spor 2 har DNA lighed med følgende person i DNA-slægtskabsdatabasen: Ejnar Kallstrøm. Ejnar Kallstrøm er pensionist og 89 år gammel.
DNA-spor 2 har DNA lighed med følgende person i DNA-slægtskabsdatabasen: Ejnar Kallstrøm. Ejnar Kallstrøm er pensionist og 89 år gammel.
Resultatet af analysen ses på kromosomkortet til højre. Identisk DNA er markeret med grønt.
Igen vurderes graden af DNA lighed. Resultatet af computeranalysen giver en lighed på ca. 12,5%.
Hvor mange generationer væk må gerningsmanden derfor findes?
Slægtskabstræet for familien ses nedenfor. Ejnar Kallstrøm er markeret med en blå firkant.

Hvilke fire mænd fra slægtskabstræet vil DNA-spor 2 kunne stamme fra? Husk at søskende antages at have 50% identisk DNA.
Da der er ca. 12,5% DNA lighed mellem Ejnar Kallstrøms DNA og DNA-spor 2, vil DNA’et fra gerningsmanden stamme fra en mand, der er 3 generationer længere væk en Ejnar Kallstrøm. Da du ved, at DNA-sporene stammer fra en mand, giver det fire mulige mistænkte: IV-6, V-12, V-13 og V-15.
Nu skal du igen bruge din viden om DNA-spor 2. Begynd med at se på dine noter. Hvilken øjenfarve og hvilken alder er blevet estimeret ud fra DNA-spor 2?
Nederst på siden ligger der en PDF med de data, som det er lykkedes politiet at indhente om de fire personer IV-6, V-12, V-13 og V-15. Læs dem igennem og brug dem til at vurdere, hvem er den mest sandsynlige gerningsmand?
 DNA-spor 3 har DNA lighed med følgende person i DNA-slægtskabsdatabasen: Gerda Lind Madsen
DNA-spor 3 har DNA lighed med følgende person i DNA-slægtskabsdatabasen: Gerda Lind Madsen
Resultatet af analysen ses igen på kromosomkortet til højre. DNA ligheden er denne gang ca. 3,2%.
Hvor mange generationer væk skal du fra Gerda Lind Madsens generation for at finde mulige gerningsmænd?
Slægtskabstræet for Gerda Lind Madsen ses nedenfor. Gerda Lind Madsen er markeret med en blå cirkel.

Baseret på DNA ligheden på ca. 3,2%, hvilke 2 personer i slægtskabstræet kan DNA-spor 3 så stamme fra?
Da der ca. er 3,2% DNA lighed mellem Gerda Lind Madsens DNA og DNA-spor 3, skal du seks generationer ud i slægtskabstræet. Da du ved, at DNA-sporene stammer fra en mand giver det to mulige mistænkte: IV-5 og IV-6.
Du skal nu bruge din viden om DNA-spor 3. Se på dine noter. Hvilken øjenfarve og hvilken alder er blevet estimeret ud fra DNA-spor 3?
Nederst på siden ligger der igen en samlet PDF med de data, som det er lykkedes politiet at indhente om de to personer IV-5 og IV-6. Vurder ud fra dem, hvem der er den mest sandsynlige gerningsmand.
Modul 8: DNA databaser og etik
- Lære om, hvad bioetik er.
- Forstå forskellen på to retninger inden for etikken – nytteetik og pligtetik.
- Se en film om, hvordan man bruger slægtskabsanalyser i retsgenetikken i USA.
- Diskutere fordele og risici ved at have store DNA-databaser.
- Finde ud af din egen holdning til den bioetiske problemstilling om DNA-databaser.
Bioteknologisk forskning åbner ofte op for en række muligheder, som vi som enkeltpersoner eller samfund bliver nødt til at forholde og til. Et eksempel på det er kloning. Vi har i dag mulighederne for at klone mennesker, så vi kan lave genetisk identiske individer af nulevende eller afdøde mennesker. Men hvad vil det betyde for den enkelte eller samfundet, hvis vi tillader det?
Sådanne spørgsmål er etiske spørgsmål. Etik handler om, hvad et godt liv er og betydningen af at tage hensyn til andre og ikke kun egne behov. Etiske spørgsmål er altså spørgsmål om, hvordan man behandler andre mennesker og andre levende organismer.
Når vi diskuterer etiske spørgsmål i forhold til bioteknologisk forskning kaldes det bioetik. Bioetikken diskuterer de problemstillinger, der opstår i forbindelse med organtransplantationer, behandlinger af sygdomme, brug af gensplejsede organismer, kloning m.m. Brug af DNA fra enkeltindivider, som du har arbejdet med i dette modul, kan altså også diskuteres bioetisk. Det skal du arbejde med nu.
Som før nævnt omhandler etikspørgsmål om, hvordan man behandler andre mennesker og andre levende organismer. Sådanne spørgsmål er ikke entydige, idet forskellige personer kan have forskellige holdninger til, hvordan det bedst gøres. Der er derfor inden for etikken forskellige retninger, som giver forslag til de bedste løsninger på etiske spørgsmål.
En retning indenfor etikken, der kan bruges til at forholde sig til bioetiske spørgsmål, er nytteetikken.
Nytteetik (utilarism på engelsk) er oprindeligt formuleret af filosoffen Jeremy Bentham i 1780’erne og videreudviklet af John Stuart Mills i 1860’erne. Nytteetikken ser på, hvordan flest mulige mennesker kan opnå størst mulig lykke og mindst mulig lidelse. Det vigtige er resultatet, konsekvensen, af de handlinger der foretages, og derfor kaldes denne form for etik også konsekvensetik.
Lad os se på et dilemma. Hvis sundhedsvæsenet har begrænsede ressourcer, er det så vigtigere, at 1000 patienter får en ny hofte og dermed bedre livskvalitet, end at 1 patient får en meget dyr behandling for en livstruende sygdom, som personen risikerer at dø af? En nytteetiker vil her argumentere for, at det, at 1000 personer får et bedre liv opvejer den store ulempe for den ene patient. Populært sagt helliger målet midlet, og den enkelte kan godt gøres til middel for flertallets lykke.
Fordi nytteetikken forholder sig til, at mængden af ressourcer i et samfund er begrænset, er det ofte den type etiske overvejelser, som politikere skal tage stilling til. I forhold til bioetik vil det f.eks dreje sig om, hvilke områder indenfor forskning, der skal støttes, hvilke behandlinger, der skal prioriteres i forhold til andre, og hvorvidt miljø skal tilgodeses mere end økonomiske interesser.
En anden etisk retning, der også er god at bruge til at forholde sig til bioetiske spørgsmål, er pligtetik.
Pligtetikken omhandler det enkelte individs rettigheder og blev formuleret af den tyske filosof Immanuel Kant i 1780’erne midt i oplysningstiden. Oplysningstiden var en tid med fokus på idealer såsom universelle menneskerettigheder. Kants vigtigste påstand var, at du skal handle sådan, at dine handlinger kan ophæves til almen lov.
Lad os se på et dilemma. Du har fået en datter, der lider af en alvorlig nyresygdom. Uden en donor, vil hun højst sandsynligt ikke overleve til sit 20 år. Din partner og dig vælger derfor at få endnu et barn, der kan donere en nyre til jeres syge barn. Er det en etisk korrekt handling?
En pligtetiker vi sige nej. Målet helliger ikke midlet. En person må altså ikke gøres til middel for din eller flertallets lykke, fordi den ene person har nogle universelle rettigheder. I det nævnte dilemma vil barn nummer to blive et middel til at sikre det syge barns lykke, men barnet skal donere en nyre og leve med konsekvenserne af det.
Du skal nu bruge din nye viden til at vurdere en etisk problemstilling. I øvelsen skal du og din gruppe diskutere, hvilke argumenter en nytteetiker eller en pligtetiker vil fremføre i forhold til følgende etisk problemstilling – Screening af fostre for Downs syndrom.
I Danmark tilbydes alle gravide en såkaldt nakkefoldsscanning i 11-14 uge af graviditeten. Hvis scanningsbilledet viser en fortykket nakkefold, som vist på figuren nedenfor, er der en øget risiko for at barnet har Downs syndrom. En ny test, non-invasiv prænatal test eller NIPT, er endnu mere præcis end nakkefoldsscanninger til at estimere risikoen for Downs syndrom, men den udføres endnu ikke som standard på hospitalerne i Danmark.

Da Downs syndrom er en kromosomfejlsmutation, hvor der er et helt eller næsten helt ekstra kromosom 21, kan en test, hvor kromosomerne kortlægges med sikkerhed fastslå, om fosteret har Downs syndrom. Sådan en test kaldes en karyotypning.

Karyotype for en pige med Downs syndrom
Hvis karyotypningen viser et ekstra kromosom 21, vil læger tage en samtale med den gravide og hendes partner og fortælle om konsekvenserne for barnet. De er listet nederst på siden. I Danmark har nakkefoldsscanningerne medført et fald i antallet af børn, der fødes med Downs syndrom, som det ses af figuren nedenfor, da mange gravide vælger at få en abort, hvis de ved, at deres kommende barn har Downs syndrom.

Udviklingen af børn født med Downs syndrom fra 1970 til 2018
Er det i orden at vælge at få en abort, hvis du ved, at dit barn kommer til at lide af Downs syndrom? Skal en læge opfordre til abort? Hvad vil en nytteetiker og en pligtetiker mene om denne etiske problemstilling? Tid til at diskutere det med din makker…
Symptomer hos personer med Downs syndrom
Følgende symptomer ses hos alle:
- Specielle fysiske træk: Rundt ansigt, skrå øjenspalter, lille næse, små ører m.m.
- Muskelslaphed og hypermobile led
- Mental retardering – dog i forskellig grad
- Væksthæmning
- Lavere gennemsnitlig levealder – gennemsnitligt 60 år men meget varierende.
Hyppigt forekommende symptomer:
- Hjertemisdannelse (ca. 45%). Kan ofte opereres.
- Nedsat stofskifte og fedmeproblemer (ca. 33 %) Kan behandles medicinsk.
- Hyppige infektioner og nedsat immunforsvar. Hvor hyppigt det forekommer er svært at sætte tal på, da det ofte er koblet til miljøet barnet vokser op i. Men det er en hyppig dødsårsag hos børn med Downs.
- Dårlig hørelse (ca. 20%) og synsproblemer (ca. 30%, øget risiko i løbet af livet i forhold til personer uden Downs)
Sjældent forekommende symptomer:
Igennem forløbet Information i DNA har i lært om, hvilke muligheder der er for at få information ud fra DNA-prøver. Men er det ønskeligt? Eller risikerer vi, at der er uønskede konsekvenser ved at indsamle store mængder DNA information i databaser.
Du skal nu se en video om, hvordan DNA slægtsskabsanalyser blev brugt til at fange en serieforbryder. I videoen rejses der nogle etiske spørgsmål, som du skal forholde dig til og diskutere med dine klassekammerater i plenum. Overvej også, hvordan en nytteetiker og en pligtetiker vil forholde sig til argumenterne i videoen.
Du har nu set videoen om DNA slægtskabsanalyser, og hvad de kan bruges og misbruges til. Du har også diskuteret videoen med dine klassekammerater. Nu er det tid til at se, hvad klassen egentlig synes. Svar på holdningsspørgsmålene nedenfor og se om dine synspunkter er på linje med flertallet i klassen.
- Vil du have lyst til at indsende dit DNA til firmaer som Ancestry, 23andMe og FamilyTreeDNA for at finde ud af hvem du er beslægtet med?
- Vil du have lyst til at indsende dit DNA til firmaer som Ancestry, 23andMe og FamilyTreeDNA for at finde ud af risikoen for at få forskellige sygdomme?
- Synes du det er et problem, at personer der uploader deres DNA til en DNA database i virkeligheden også uploader de dele af deres slægtninges DNA, som de deler med hinanden?
Skal politiet have adgang til DNA databaser fra firmaer som Ancestry, 23andMe og FamilyTreeDNA, så de kan løse sager som Golden State Killer sagen, også selvom personer, der har indsendt DNA, ikke har givet tilladelse til det?
Video venligst stillet til rådighed af Nationalt Genom Centers fra deres hjemmeside: https://ngc.dk
Siden 2019 har vi i Danmark haft et Nationalt Genom Center (NGC), der er en styrelse under Sundhedsministeriet. NGC’s opgave er at indsamle data fra omfattende genetiske analyser og stille dem til rådighed for læger og forskere for at forbedre udredning, diagnosticering og behandling af udvalgte patientgrupper samt at drive Danmarks nationale infrastruktur for personlig medicin.
Personlig medicin er medicin, der gives på baggrund af genetiske undersøgelser, så læger får mere præcise diagnoser at behandle ud fra. Udviklingen af personlige medicinske behandlinger er i gang med at revolutionere en række behandlinger, herunder behandlingen af en lang række typer af kræft, der ellers har været svære at behandle.
NGC har et supercomputernetværk, der skal muliggøre analysen af de indsamlede data. Patienter, der skal have udført en genetisk undersøgelse, skal før analysen give skriftligt samtykke.
NGC_samtykkeblanket til omfattende genetisk analyse i behandling_10.12.2020 Dine genetiske oplysninger, dit valg_28.09.2021
Data skal ifølge loven indberettes til og opbevares i NGC, men ved at tilmelde sig vævsanvendelsesregistret kan du vælge, at data ikke benyttes til forskning.
Dine genetiske oplysninger, dit valg_28.09.2021
NGC har desuden omfattende sikkerhedsforanstaltninger, hvad angår informationssikkerhed og persondatabeskyttelse.
Du har i temaet Information i DNA lært om, hvilke informationer om en person, der kan udledes vha. DNA-analyser. Du skal nu tage stilling til et nyt dilemma. Du har fået foretaget en genetisk undersøgelse pga. en sygdom og skal nu tage stilling til, om du vil lade data fra dit DNA indgå i fremtidige forskningsprojekter. Hvordan kan du argumentere hhv. for og imod?
I første modul blev du bedt om at lave en brainstorm og skrive ned, hvad du vidste, at DNA kunne fortælle om dig, og hvad du troede, at DNA kunne fortælle om dig.
Du er nu færdig med temaet ‘Information i DNA’. Hvor godt passede dine forudsigelser? Og er der noget, der har overrasket dig i forhold til, hvad DNA kan fortælle om dig?